单细胞细胞通讯全流程分析教程,代做分析和辅导
0. 分析参数文件和细胞通讯的演示数据
0.1 细胞通讯分析总的参数文件,后面部分细胞通讯分析模块会用到
分析参数文件
参数文件名称:total_analysis_params_demo.xlsx ,很多分析模块都是这个总的参数文件,我的这个总的参数文件如果有更新的话,我就上传到百度网盘。包括拟时序分析的所有分析模块都是用的这一个参数文件
从百度网盘下载该参数文件,该参数文件在百度网盘的位置:
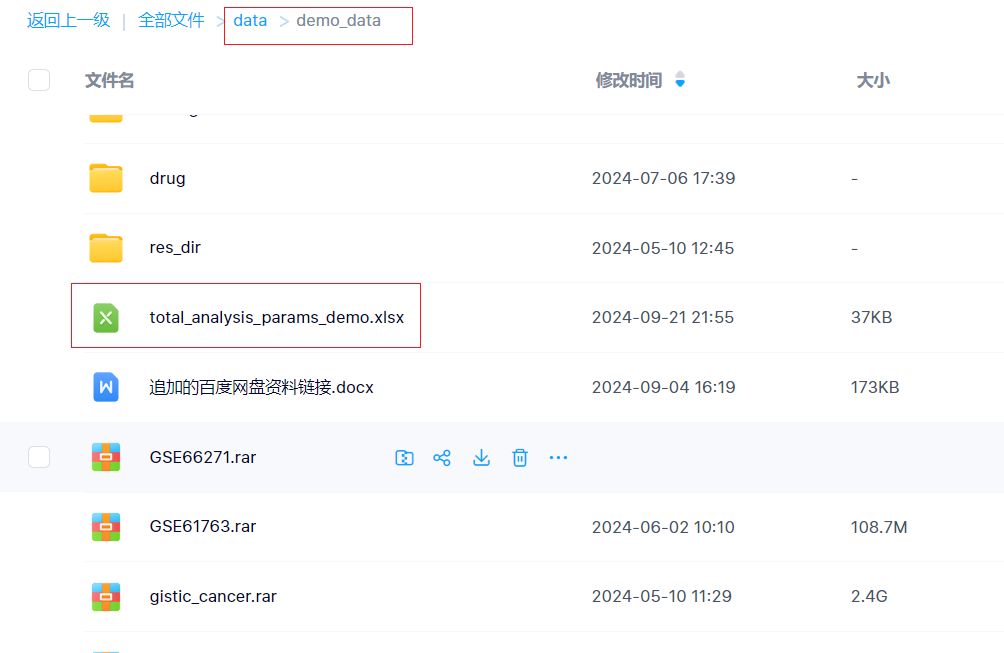
我们可以把这个参数文件下载到本地的:D:/omics_tools/demo_data这个目录下。下载后,在本地的文件为:
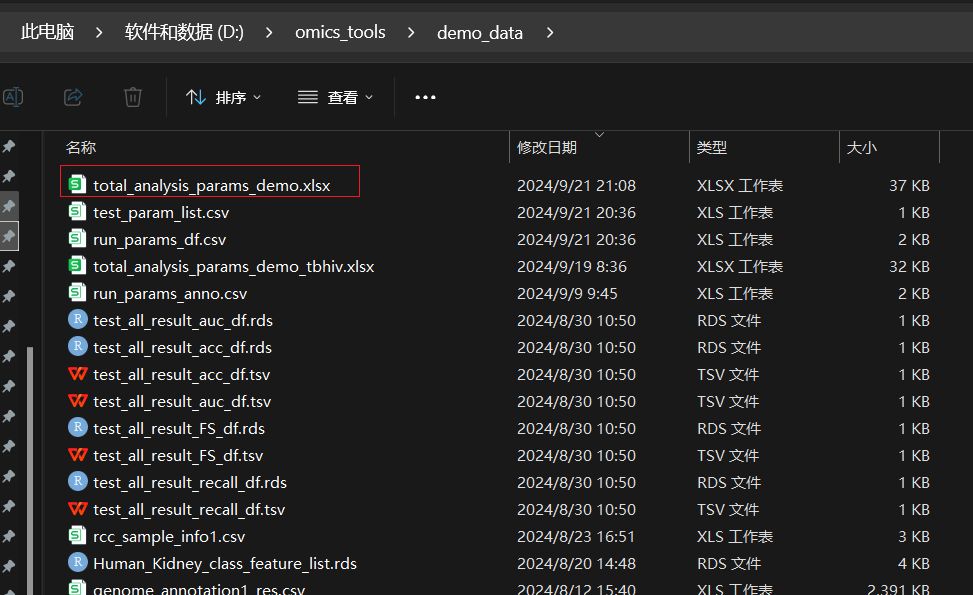
拟时序分析所有模块都用的这个总的参数文件的完整的文件路径就是:D:/omics_tools/demo_data/total_analysis_params_demo.xlsx
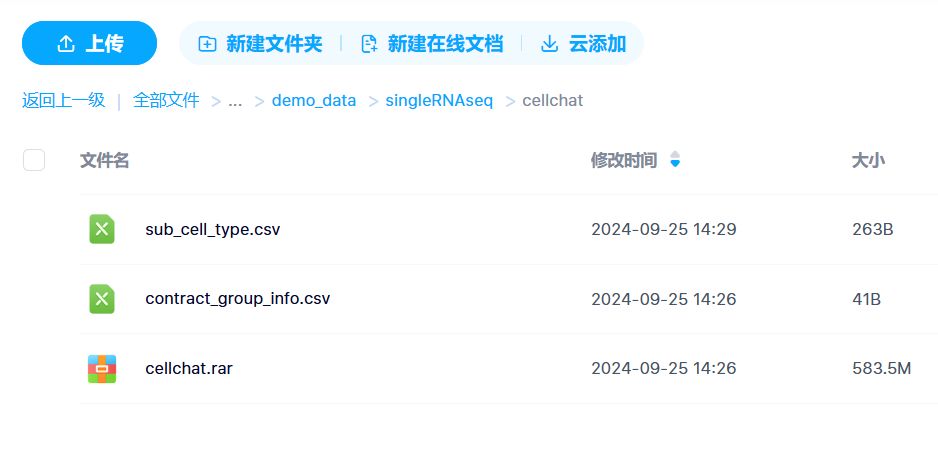
细胞通讯分析的演示数据集
0.2 分析思路
0.3 每个模块下的教学视频
每个分析模块下都放了一个对应的b站教学视频链接,大家都可以看该模块下对应的b站教学视频来学习该模块的使用方法
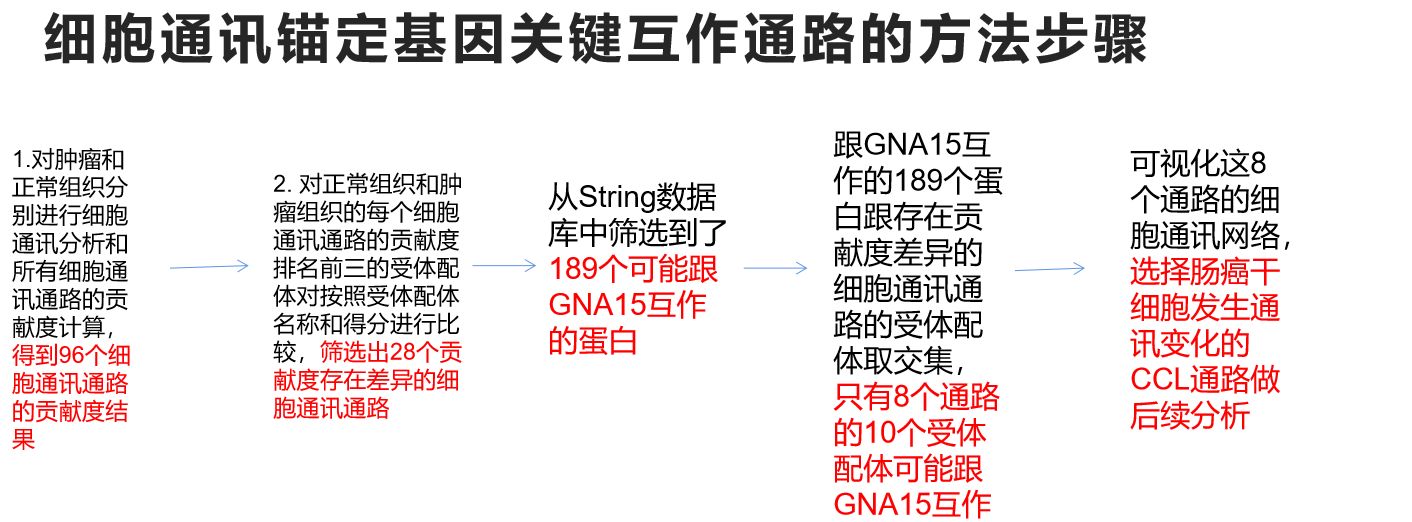
1.细胞通讯分数计算和差异贡献度通路筛选
1.1对每个分组单独进行所有类型细胞的细胞通讯分析计算
分析模块位置
软件界面

使用我OmicsTools前面单细胞的seurat系列分析注释好细胞名称的rds文件来做这一步的细胞通讯分析,这一步分析耗时比较长,可能会要运行1-2个小时。
运行完成的结果
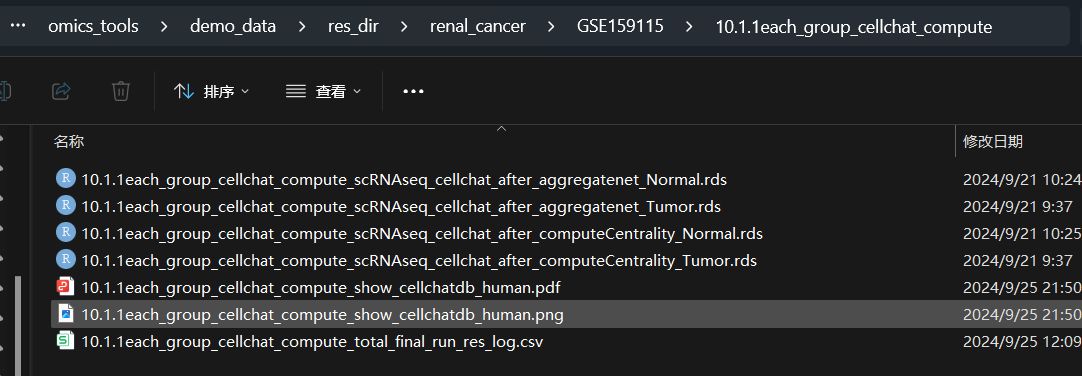
这里我用了Tumor和Normal两个分组,每个分组都使用CellChat计算处理细胞通讯网络的两个rds文件,后续的分析需要用到这一步的结果时,直接给出这里细胞通讯分析的这个结果目录的目录路径就可以了。
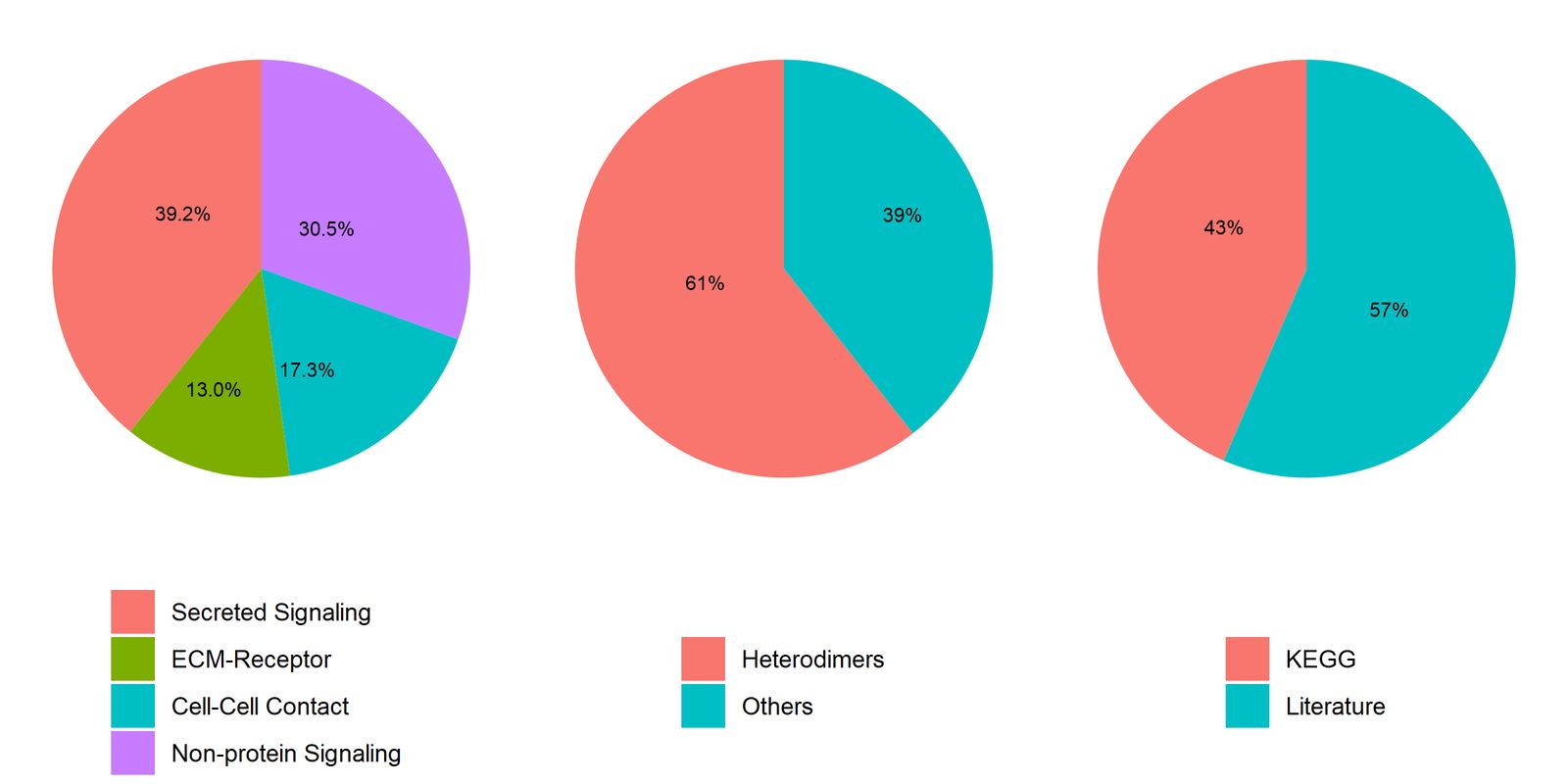
这是CellChat细胞通讯数据库的信号通路类别占比百分比的汇总图,展示了每种类别的包含的这些信号通路的百分比情况。
1.2对部分感兴趣细胞的细胞通讯分析计算(这一步可以放到后面再做,先把总的细胞通讯做的差不多了,找到感兴趣的细胞再做这一步)
软件界面

使用我OmicsTools前面单细胞的seurat系列分析注释好细胞名称的rds文件同时给出一组感兴趣的细胞名称来做这一步的细胞通讯分析,这一步分析耗时比较长,可能会要运行1-2个小时。
一组感兴趣的细胞名称的示例文件sub_cell_type.csv内容如下:
运行完成的结果
这里我用了Tumor和Normal两个分组和一组感兴趣的细胞,每个分组都使用CellChat计算处理特定的一组细胞间的细胞通讯网络的两个rds文件,后续的分析需要用到这一步的结果时,直接给出这里细胞通讯分析的这个结果目录的目录路径就可以了。
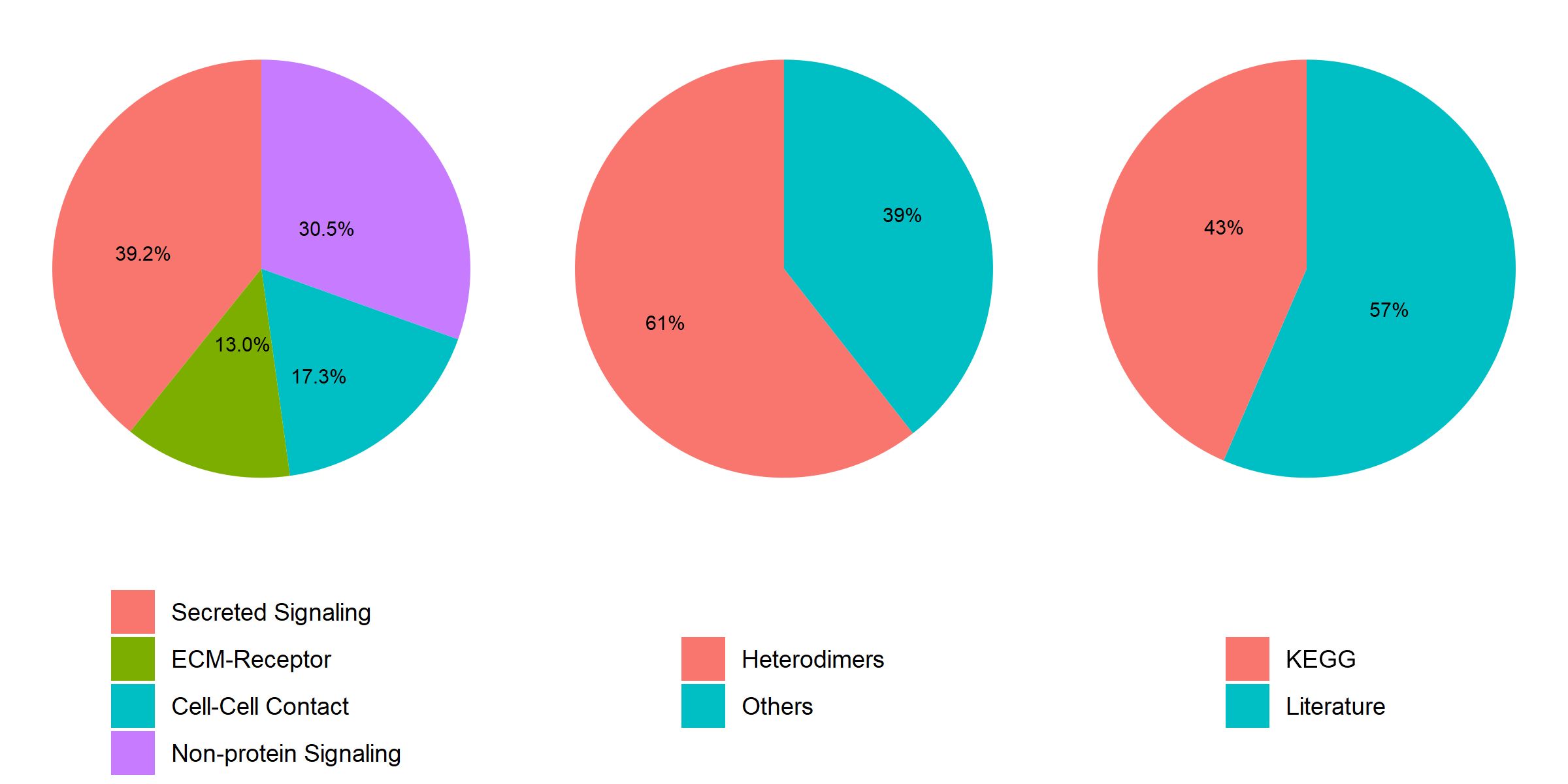
这是CellChat细胞通讯数据库的信号通路类别占比百分比的汇总图,展示了每种类别的包含的这些信号通路的百分比情况。
1.3对每个分组总的细胞通讯可视化和lr贡献度可视化
软件界面
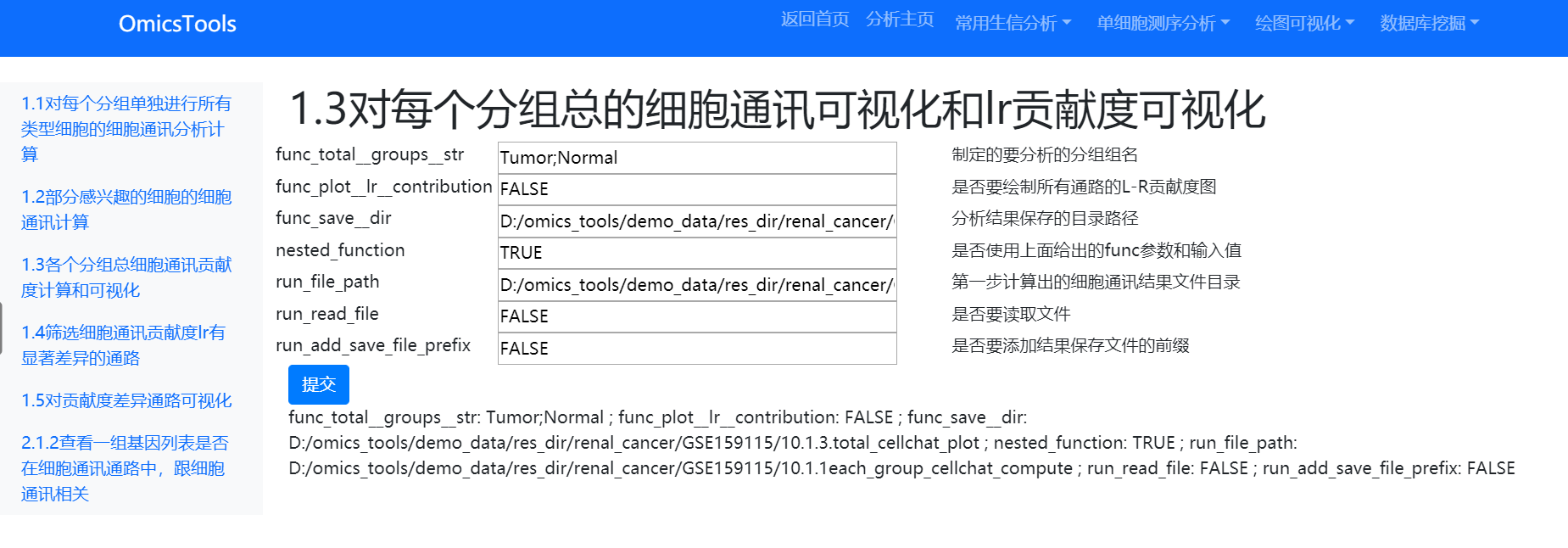
该步的分析结果目录主要是存放在func_save_dir指定的目录路径下
分析结果
结果文件列表
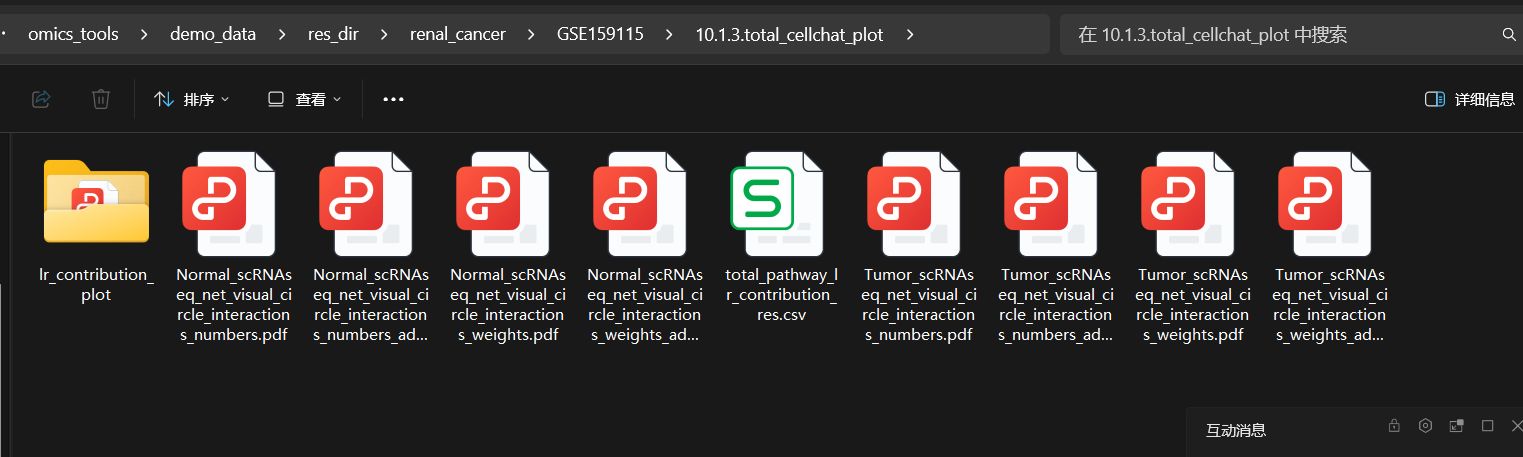
细胞通讯网络的介绍
CellChat是一个用于分析和可视化细胞间通讯网络的工具。在CellChat的输出中,interaction numbers和interaction weights是两个重要的指标,它们分别代表:
1. Interaction numbers (交互数量):
这个指标表示在细胞间通讯网络中检测到的显著交互的数量。具体来说:
- 它反映了不同细胞类型之间可能发生的配体-受体对的数量。
- 每一个检测到的配体-受体对都被计为一个交互。
- 较高的交互数量可能意味着两种细胞类型之间有更多样化的通讯方式。
2. Interaction weights (交互权重):
这个指标衡量了细胞间交互的强度或重要性。具体含义是:
- 它通常基于配体和受体的表达水平来计算。
- 权重值越高,表示该交互在细胞通讯网络中可能越重要。
- 它考虑了细胞数量、基因表达水平等因素,给出了一个综合的交互强度评分。
这两个指标结合起来可以帮助研究者更全面地理解细胞间通讯的模式:
- Interaction numbers告诉我们通讯的配体-受体对的数量
- Interaction weights则反映了这些通讯的相对重要性
在分析CellChat结果时,通常需要同时考虑这两个指标,以获得细胞间通讯网络的完整图景。
这两个指标的数值范围及其含义:
1. Interaction numbers (交互数量):
数值范围:通常是非负整数,从0开始,上限取决于数据集的特性。
- 0:表示两种细胞类型之间没有检测到显著的交互。
- 1-5:较低的交互数量,可能表示细胞类型之间的通讯有限。
- 6-20:中等水平的交互,表示存在多种通讯途径。
- 20以上:高水平的交互,表明细胞类型之间有丰富的通讯网络。
具体的数值范围可能因研究而异,取决于研究的规模、细胞类型的多样性以及使用的配体-受体数据库。
2. Interaction weights (交互权重):
数值范围:通常是非负实数,CellChat中常见的范围可能在0到1之间,但也可能超过1,具体取决于权重的计算方法。
- 0-0.2:弱交互,可能不太重要或不太可靠。
- 0.2-0.5:中等强度的交互,可能有一定的生物学意义。
- 0.5-0.8:较强的交互,在细胞通讯网络中可能扮演重要角色。
- 0.8-1.0或更高:非常强的交互,可能是关键的细胞通讯途径。
没有标记通讯交互数量或通讯强度的细胞通讯总图
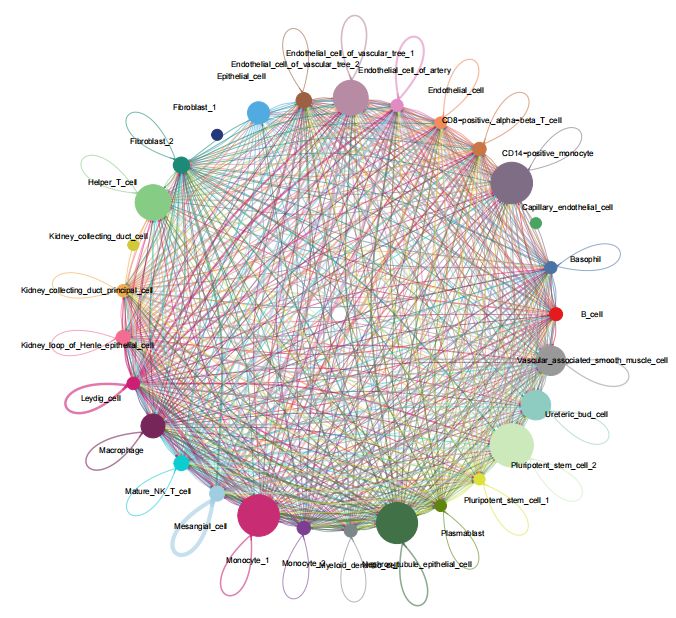
标记了细胞通讯交互数量的图

标记了细胞通讯交互强度的图
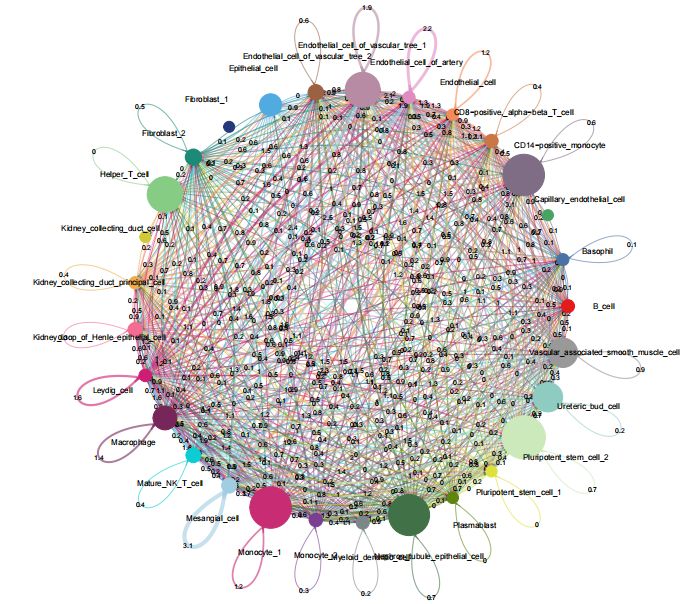
形成闭环的线是细胞的自分泌通讯,没有形成闭环的线是细胞间的旁分泌通讯。
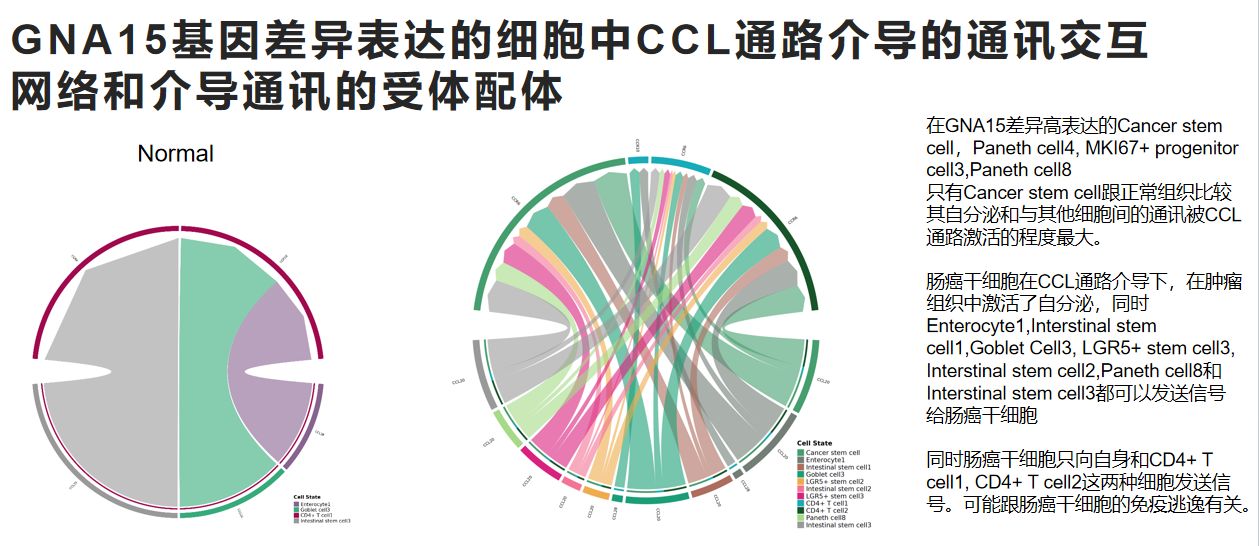
将待研究的基因跟细胞通讯网络联系起来的结果解读,以CABLES1基因为例
CABLES1基因在肾癌的肿瘤vs正常组织中的单细胞所有细胞类型中的差异表达结果
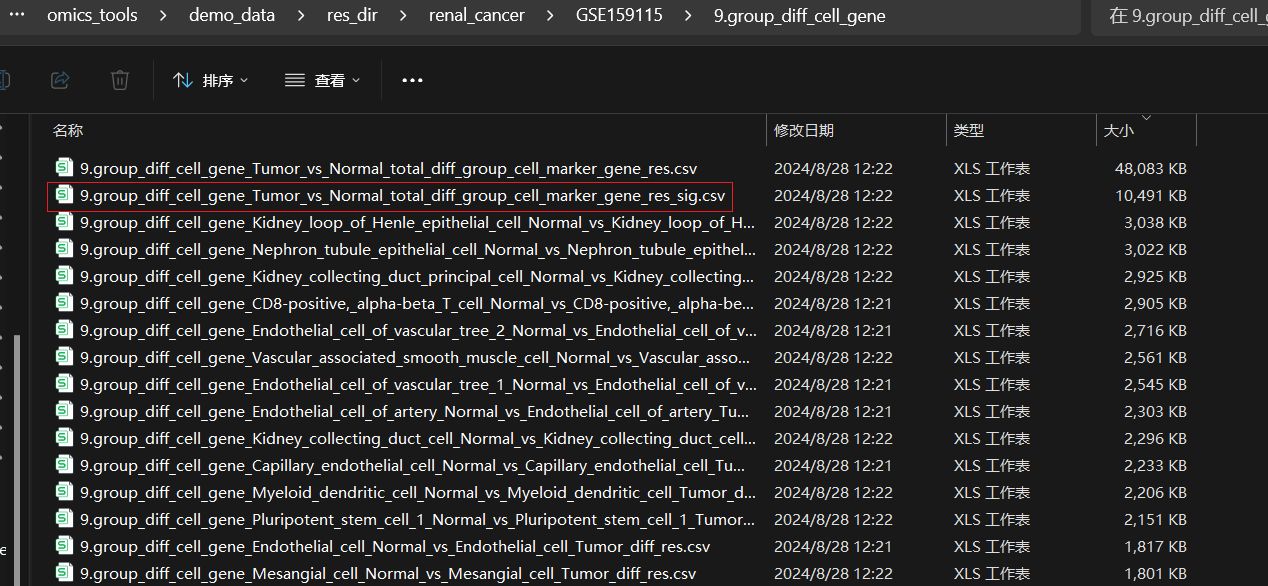
查看使用seurat之前做的差异分析结果中有该基因表达差异的细胞名称跟细胞通讯的通讯互作网络联合起来解读
1.4 根据通路中受配体的lr贡献度得分提取出贡献度显著差异的通路
软件界面

运行结果
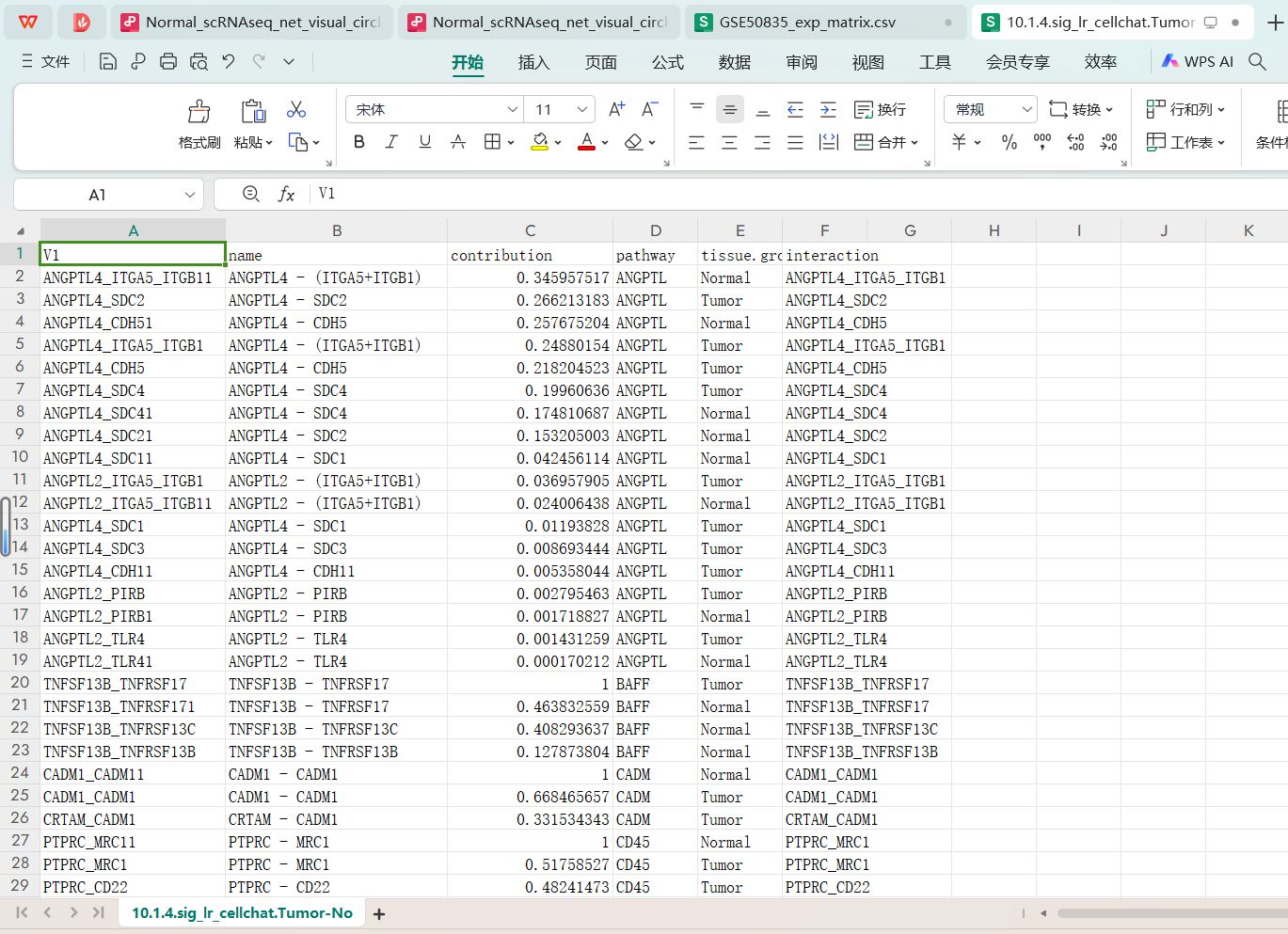
10.1.4.sig_lr_cellchat.Tumor-Normal.sig_pathway_lr_contribution_res.csv该文件是有差异的通路的汇总文件,这些差异的通讯通路一般是细胞通讯中的贡献度排名最高的受体-配体对在正常或肿瘤间改变了名字变成了其它的受体-配体对或者二者的值差距较大。
1.5对贡献度差异通路可视化
软件界面

运行结果
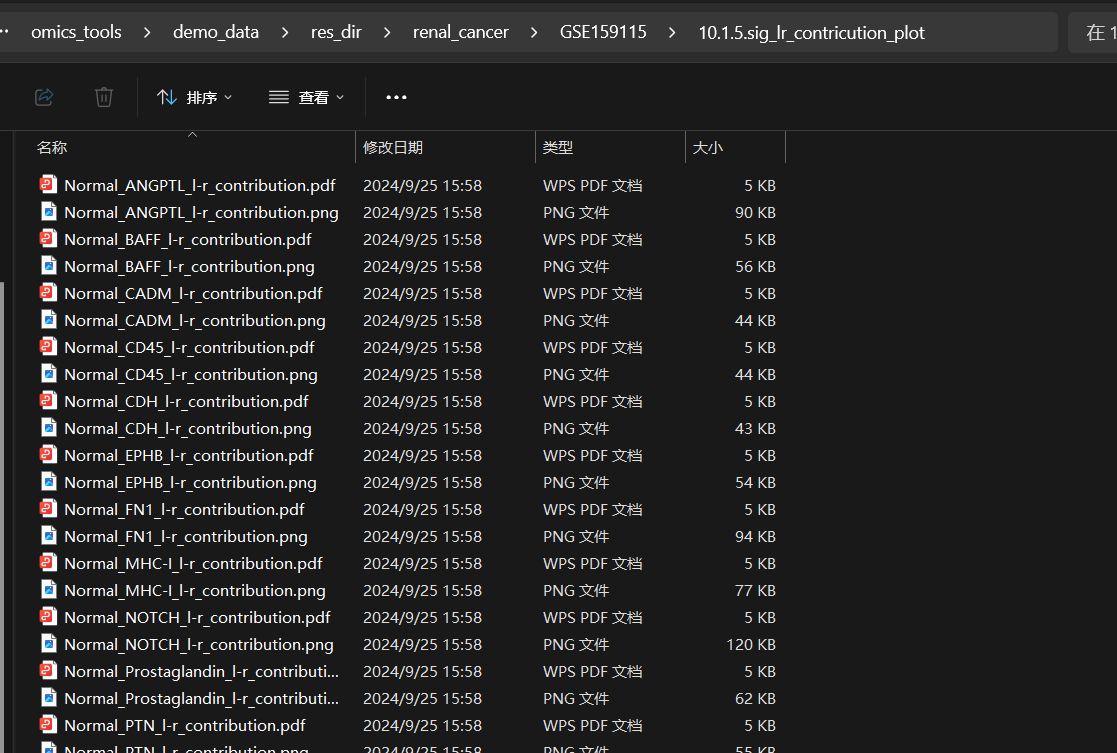
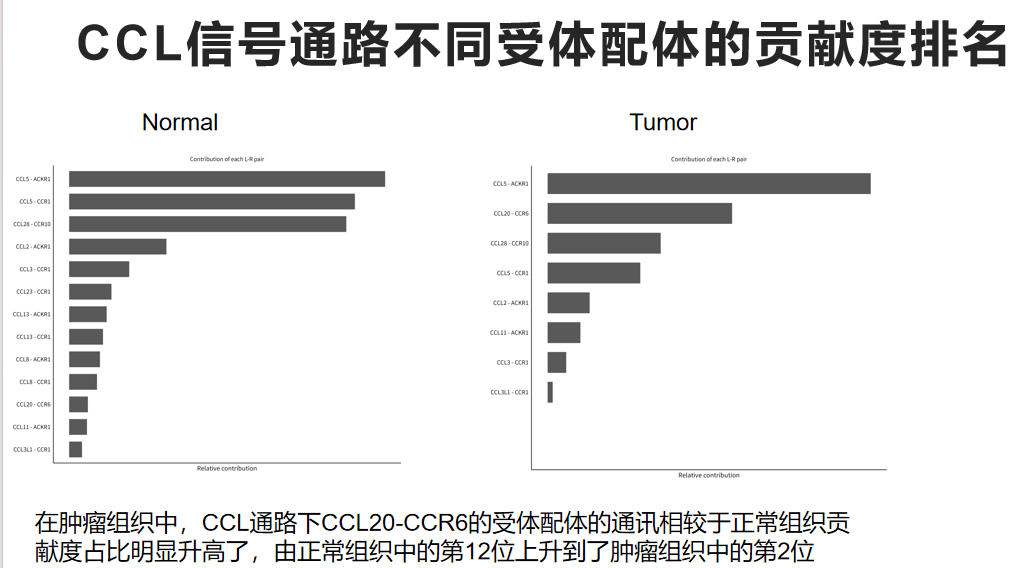
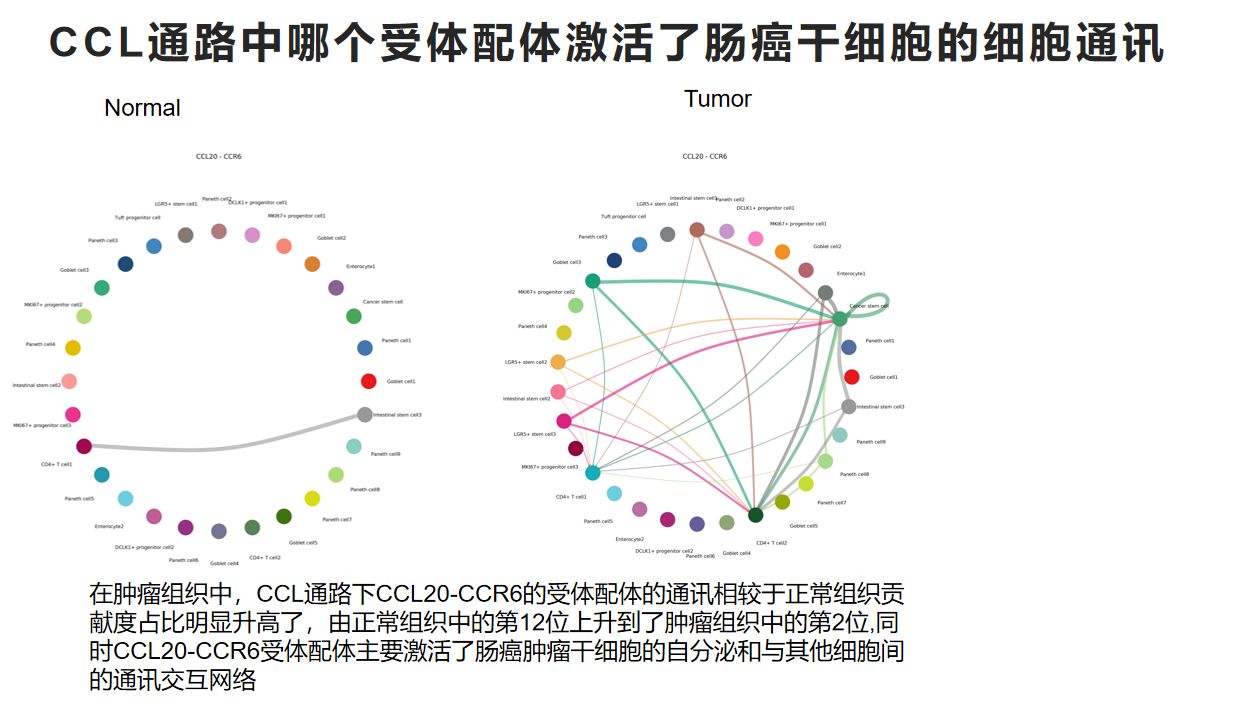
Nomal中的CCL通路
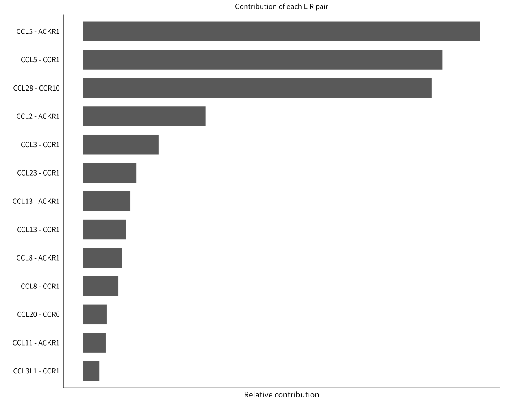
Tumor中的CCL通路
在肿瘤组织中,CCL通路下CCL20-CCR6的受体配体的通讯相较于正常组织贡献度占比明显升高了,由正常组织中的第12位上升到了肿瘤组织中的第2位
对要研究的基因互作的通讯通路受体配体对和细胞进行筛选和可视化
2.1.1 获取某个基因的核心ppi互作蛋白列表
软件界面
运行结果
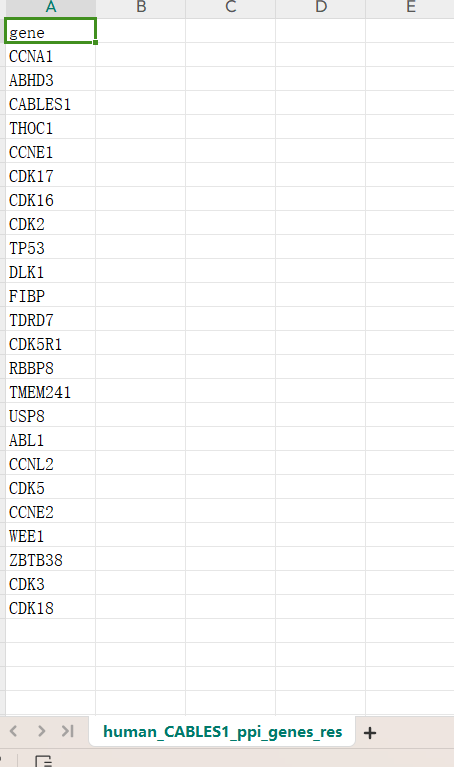
得到了string数据库中跟我们想研究的CABLES1的一组互作基因列表
2.1.2查看一组基因列表是否在细胞通讯通路中,跟细胞通讯相关
软件界面
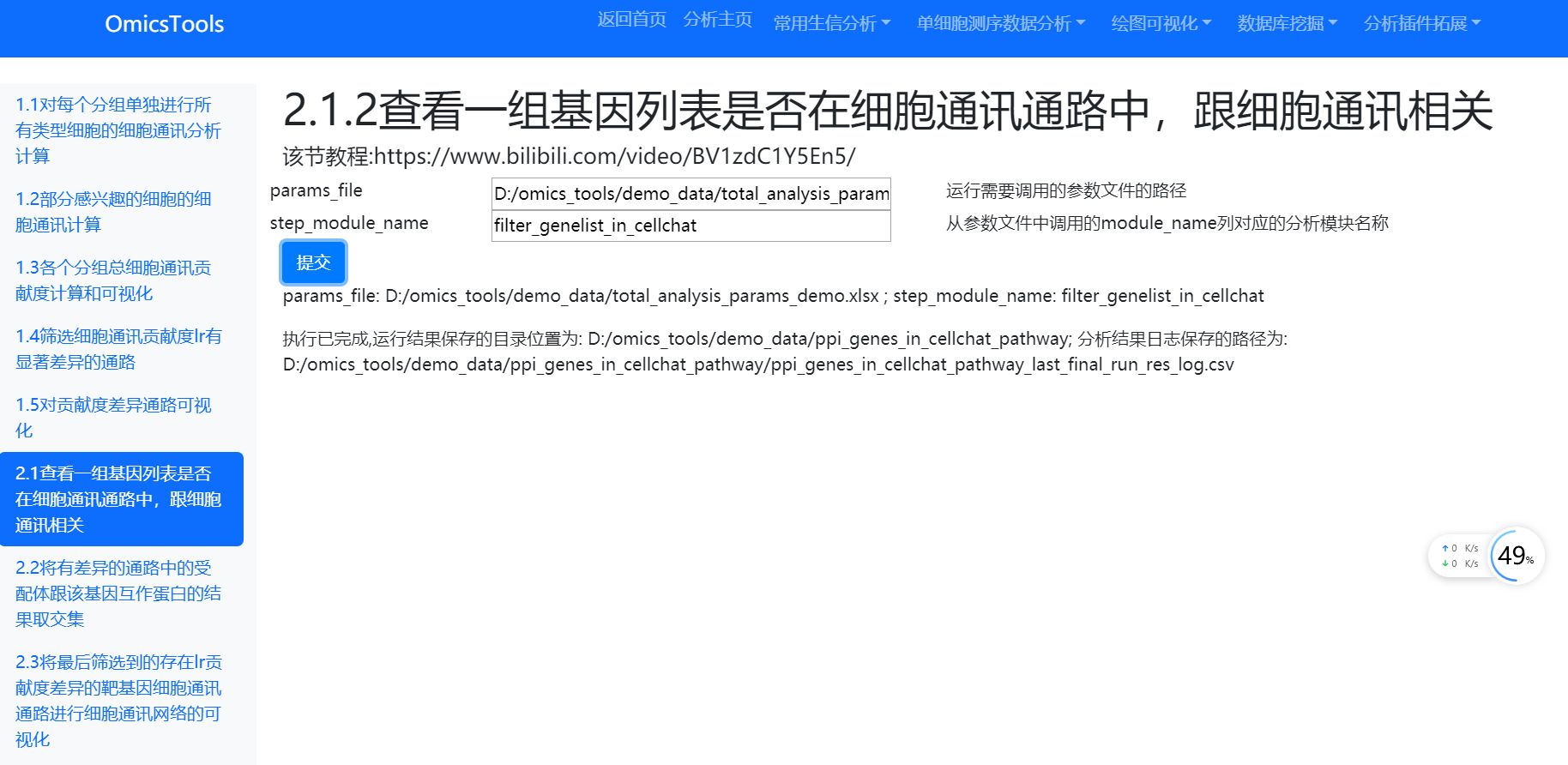
在excel中对分析参数进行修改
该分析模块的名称step_module_name是filter_genelist_in_cellchat,即用的是这个总的参数文件的完整的文件:D:/omics_tools/demo_data/total_analysis_params_demo.xlsx 里面的该模块的参数值。
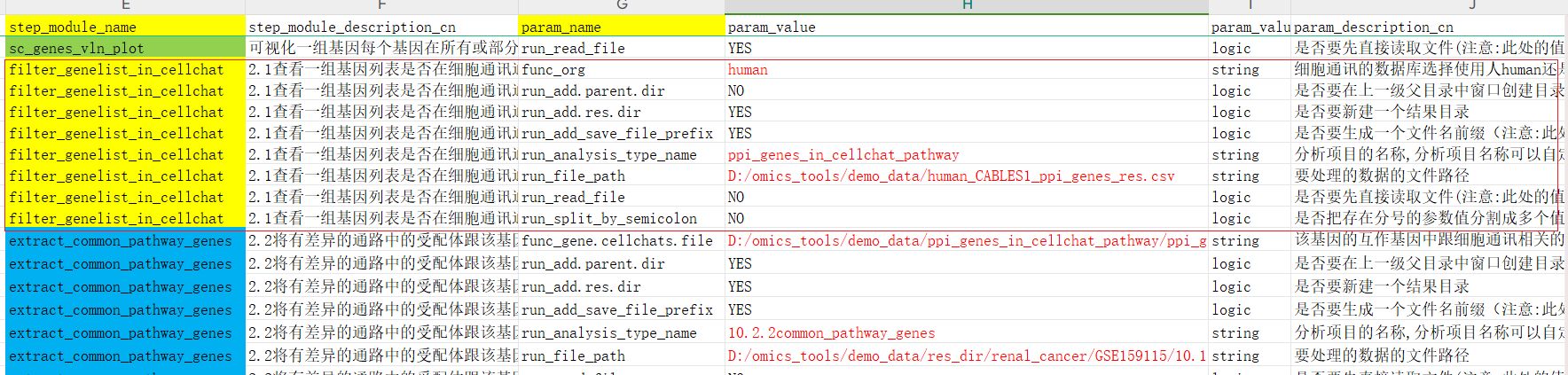
修改参数值的时候,只用改这个模块的参数值中我标红的那几个参数值,包括大家电脑中要分析的数据的文件路径和一些要调整和特别指定的重要参数值,没有标红的参数值,大家一律用默认的,不用修改。
运行结果
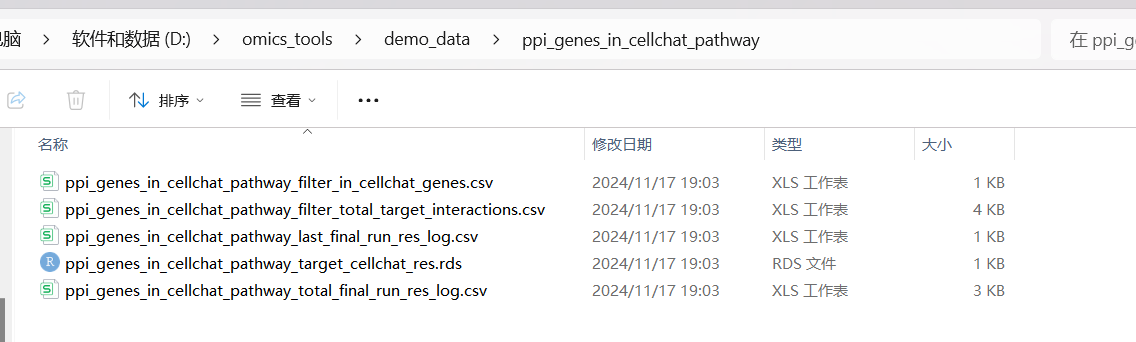
这个里面是用的CABLES1的一些互作蛋白的基因名称列表跟CellChat细胞通讯数据库中的通路中的受体配体列表取交集,得到跟我们想研究的基因的互作蛋白哪些也是属于细胞通讯通路中的受体配体的结果表格文件。
ppi_genes_in_cellchat_pathway_filter_in_cellchat_genes.csv文件中的结果:
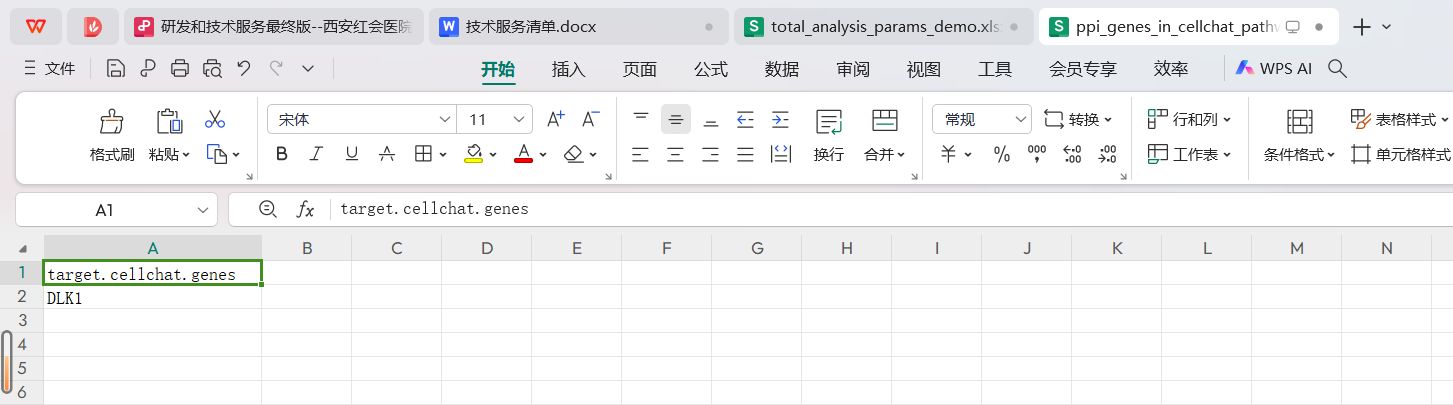
只找到了在细胞通讯通路受体配体中只有一个基因是CABLES1的互作蛋白DLK1。
ppi_genes_in_cellchat_pathway_filter_total_target_interactions.csv文件中的结果:
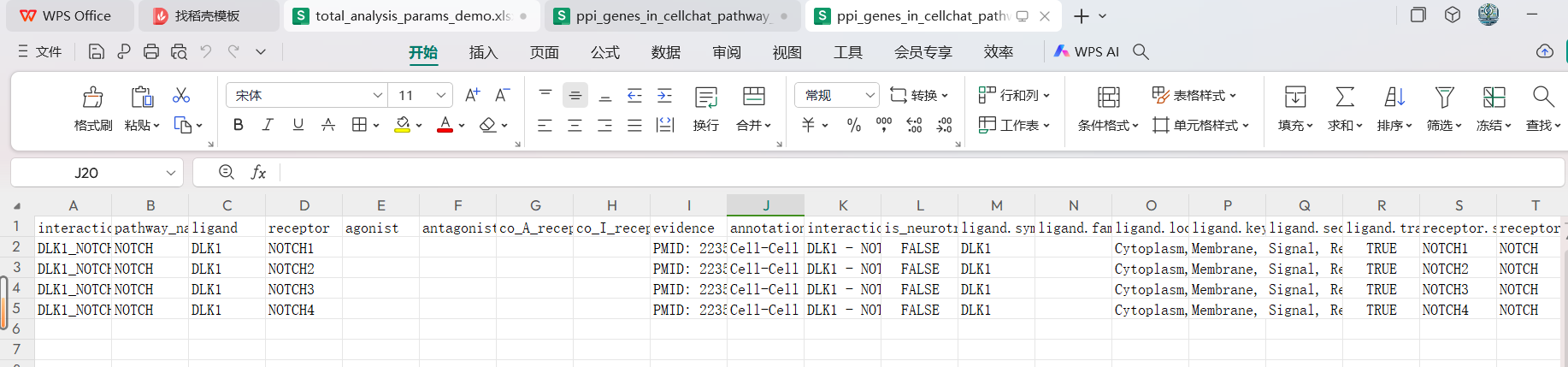
这是DLK1配体所属的NOTCH通路以及该通路中跟DLK1互作通讯的受体配体对情况
2.2将有差异的通路中的受配体跟该基因互作蛋白的结果取交集
软件界面
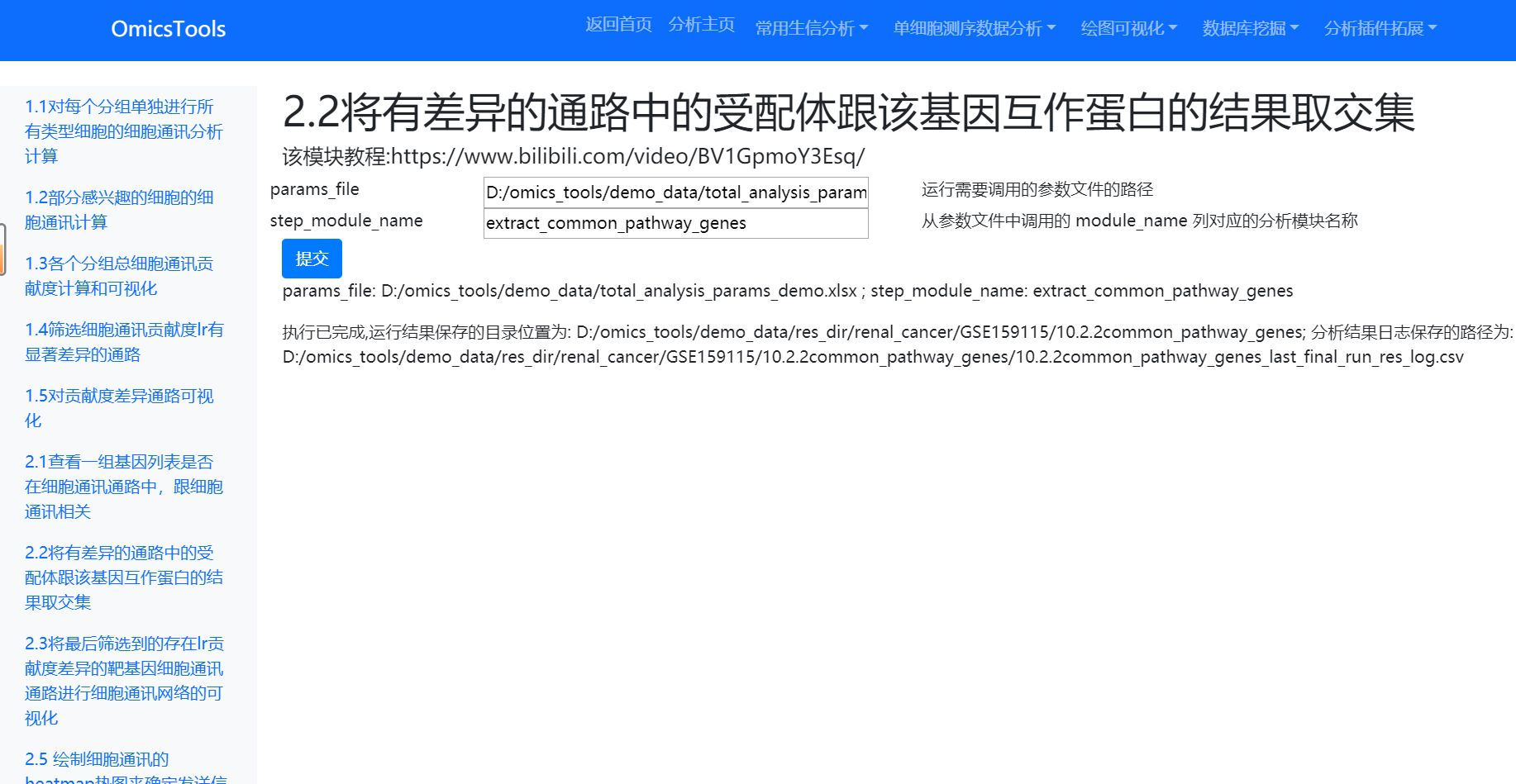
在excel中对分析参数进行修改
该分析模块的名称step_module_name是extract_common_pathway_genes,即用的是这个总的参数文件的完整的文件:D:/omics_tools/demo_data/total_analysis_params_demo.xlsx 里面的该模块的参数值。

修改参数值的时候,只用改这个模块的参数值中我标红的那几个参数值,包括大家电脑中要分析的数据的文件路径和一些要调整和特别指定的重要参数值,没有标红的参数值,大家一律用默认的,不用修改。
运行结果
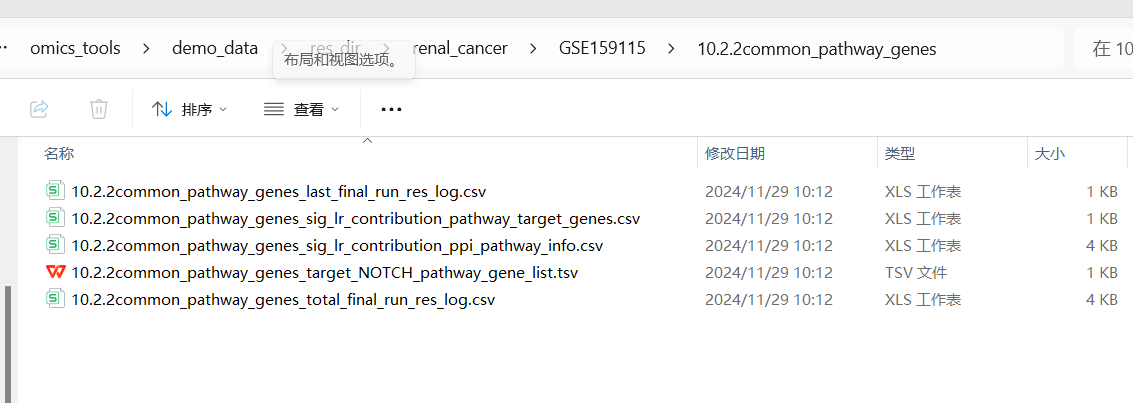
这是将有差异的通路中的受配体跟该基因互作蛋白的结果取交集的结果文件列表
10.2.2common_pathway_genes_sig_lr_contribution_pathway_target_genes.csv文件中的内容:
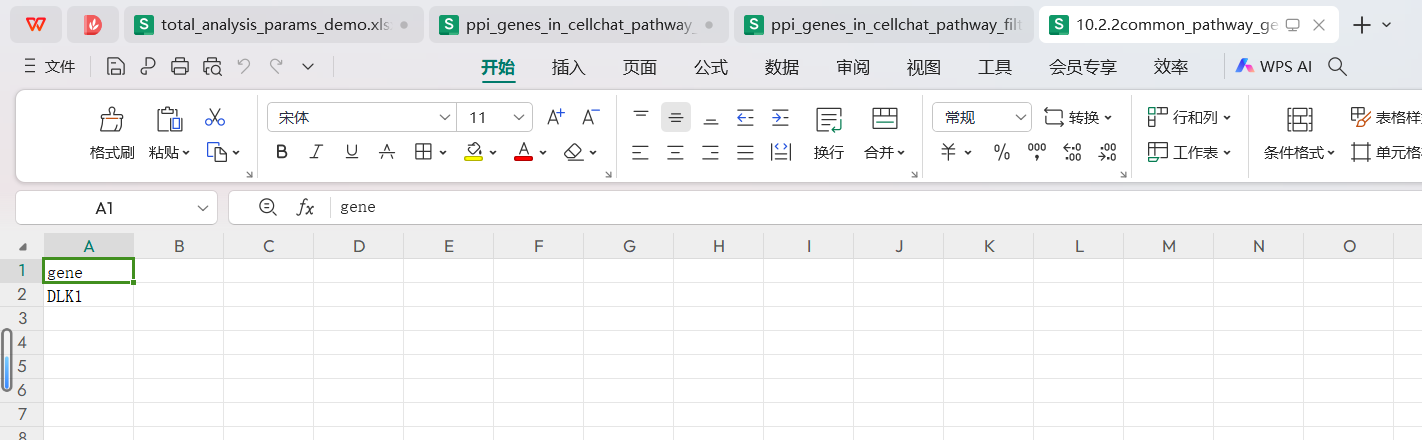
只有DLK1这一个基因既是CABLES1的互作蛋白,又是细胞通讯贡献度有差异通路中的受体配体对。
10.2.2common_pathway_genes_sig_lr_contribution_ppi_pathway_info.csv文件中的内容:

这是交集中贡献度有差异的NOTCH通路以及该通路中跟DLK1互作通讯的受体配体对详细信息
10.2.2common_pathway_genes_target_NOTCH_pathway_gene_list.tsv文件中的内容:
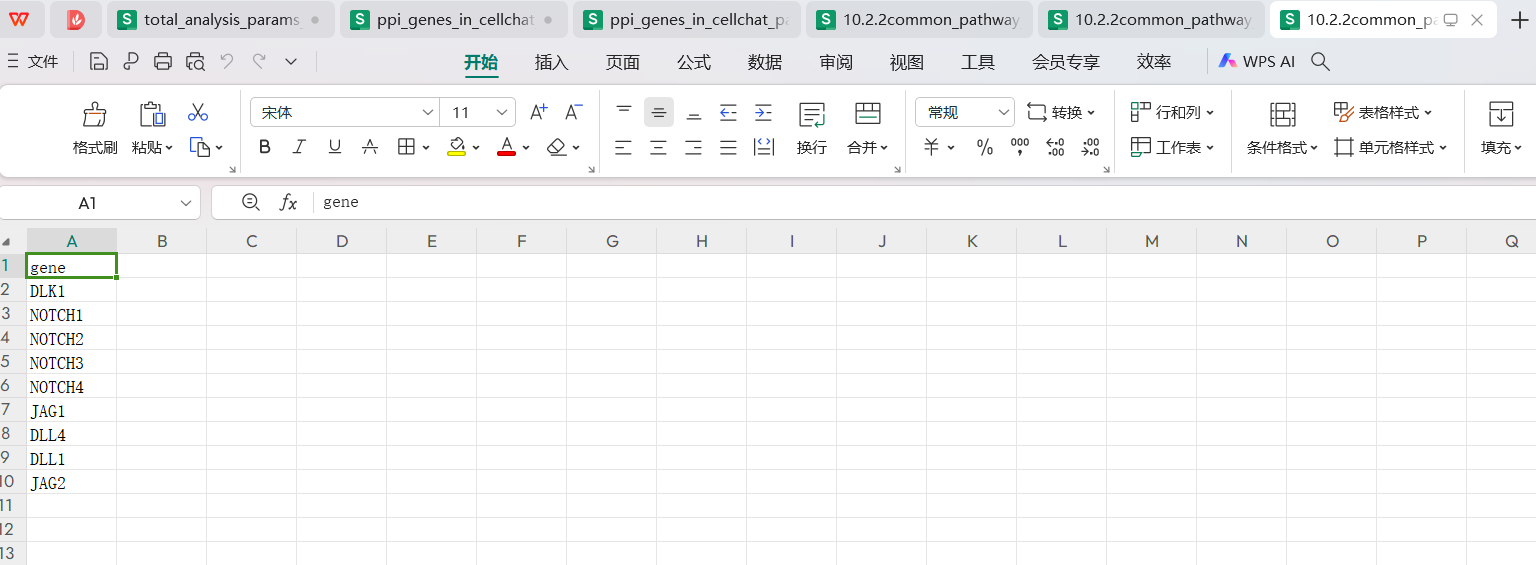
这是交集中贡献度有差异的NOTCH通路以及该通路中跟DLK1互作通讯的受体配体对详细基因列表
2.3将最后筛选到的存在lr贡献度差异的靶基因细胞通讯通路进行细胞通讯网络的可视化
软件界面
在excel中对分析参数进行修改
该分析模块的名称step_module_name是sig_lr_pathway_cellchat_plot,即用的是这个总的参数文件的完整的文件:D:/omics_tools/demo_data/total_analysis_params_demo.xlsx 里面的该模块的参数值。
修改参数值的时候,只用改这个模块的参数值中我标红的那几个参数值,包括大家电脑中要分析的数据的文件路径和一些要调整和特别指定的重要参数值,没有标红的参数值,大家一律用默认的,不用修改。
运行结果
结果文件列表
这些图都是肿瘤组和正常组都是贡献度条形图barplot,通讯网络circle图,通讯网络的和弦图chord图各画了一个图,在解读图片结果的时候,需要把正常组织的结果图和肿瘤组织的结果图对比着来看。
贡献度图比较分析
通讯网络circle图,通讯网络的和弦图chord图比较分析
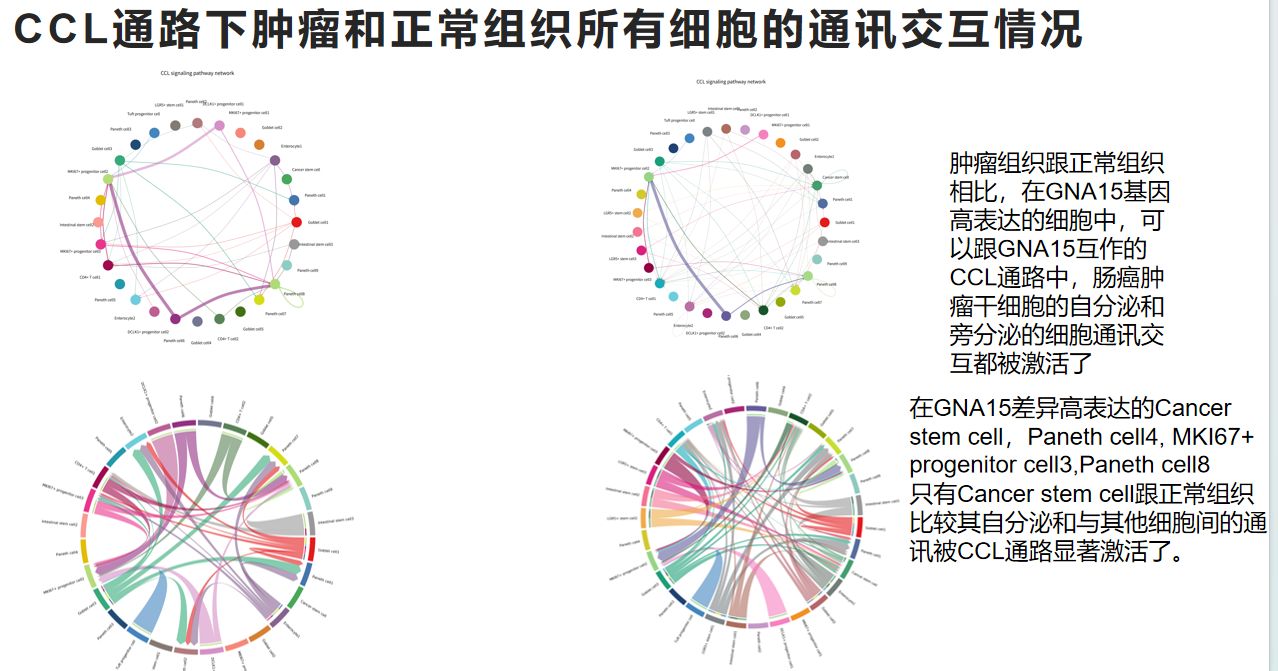
2.4 感兴趣的基因的互作蛋白与筛选到的细胞通讯通路的受体配体的ppi互作网络
把2.2步将有差异的通路中的受配体跟该基因互作蛋白的结果取交集基因列表放到String数据库网站中就可以得到这样到互作网络图了
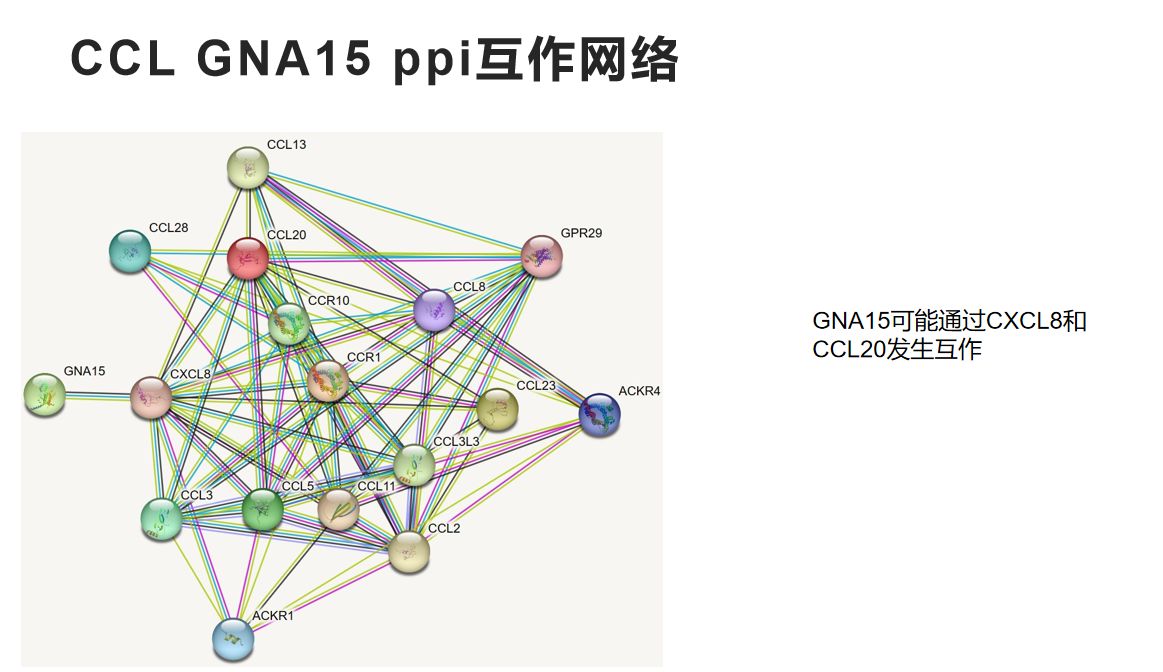
2.5 绘制细胞通讯的heatmap热图来确定发送信号和接受信号的重要细胞
软件界面
在excel中对分析参数进行修改
该分析模块的名称step_module_name是cellchat_heatmap_plot,即用的是这个总的参数文件的完整的文件:D:/omics_tools/demo_data/total_analysis_params_demo.xlsx 里面的该模块的参数值。
修改参数值的时候,只用改这个模块的参数值中我标红的那几个参数值,包括大家电脑中要分析的数据的文件路径和一些要调整和特别指定的重要参数值,没有标红的参数值,大家一律用默认的,不用修改。
运行完成的结果
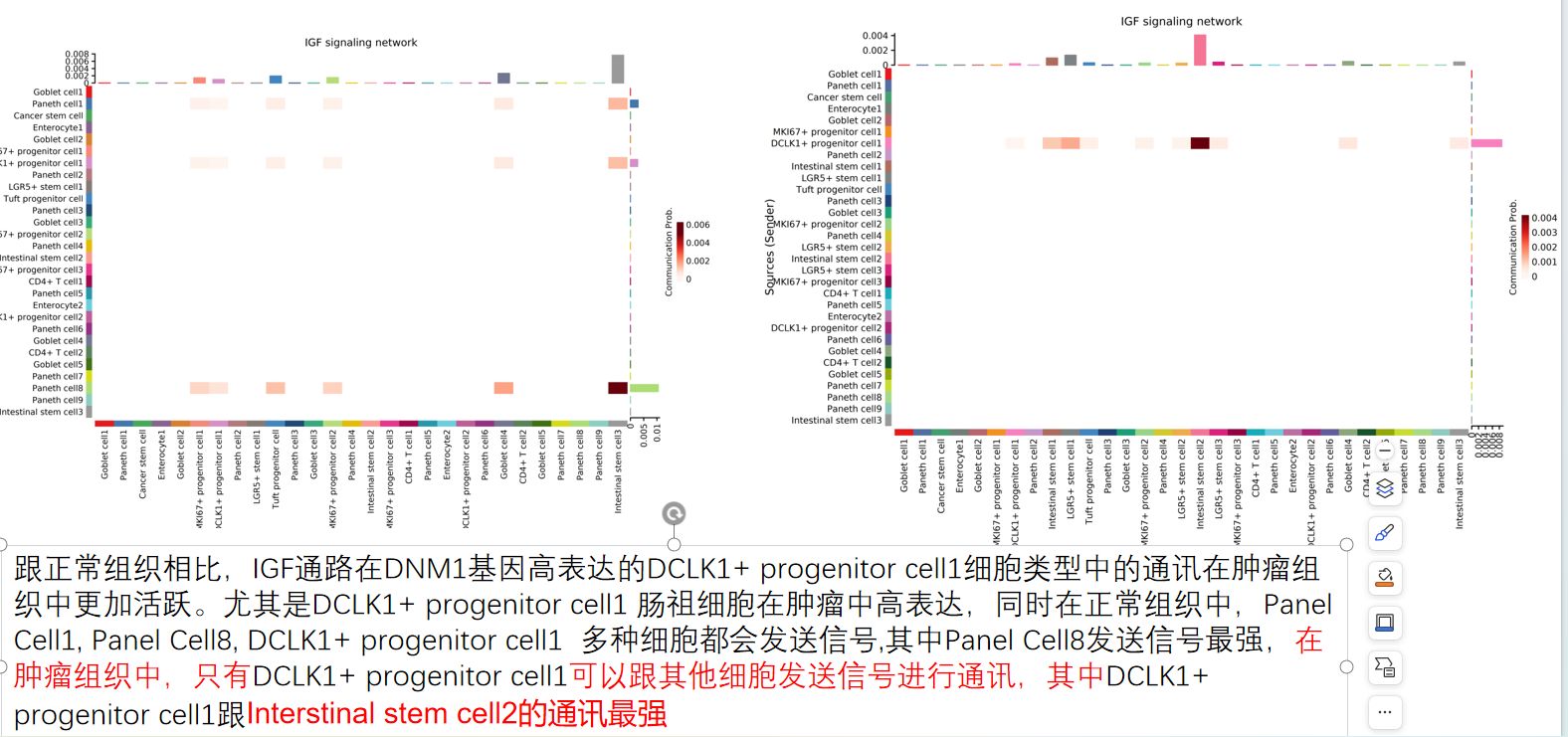
把Normal和Tumor的热图像这样放在一起比较
2.6 对筛选好的细胞通讯通路和感兴趣细胞的细胞通讯进行详细的可视化分析
软件界面

在excel中对分析参数进行修改
该分析模块的名称step_module_name是sig_pathway_cellchat_show,即用的是这个总的参数文件的完整的文件:D:/omics_tools/demo_data/total_analysis_params_demo.xlsx 里面的该模块的参数值。
修改参数值的时候,只用改这个模块的参数值中我标红的那几个参数值,包括大家电脑中要分析的数据的文件路径和一些要调整和特别指定的重要参数值,没有标红的参数值,大家一律用默认的,不用修改。
运行完成的结果
结果文件列表
这一步会得到特别多的图
该模块结果中的子步骤cellchat1的结果图
该通路在肿瘤和正常组织中的细胞通讯网络图
该模块结果中的子步骤cellchat2的结果图
该模块结果中的子步骤cellchat3的结果图
该通路下每个受体-配体对介导的细胞通讯网络
该模块结果中的子步骤cellchat4的结果图
该模块结果中的子步骤cellchat5的结果图
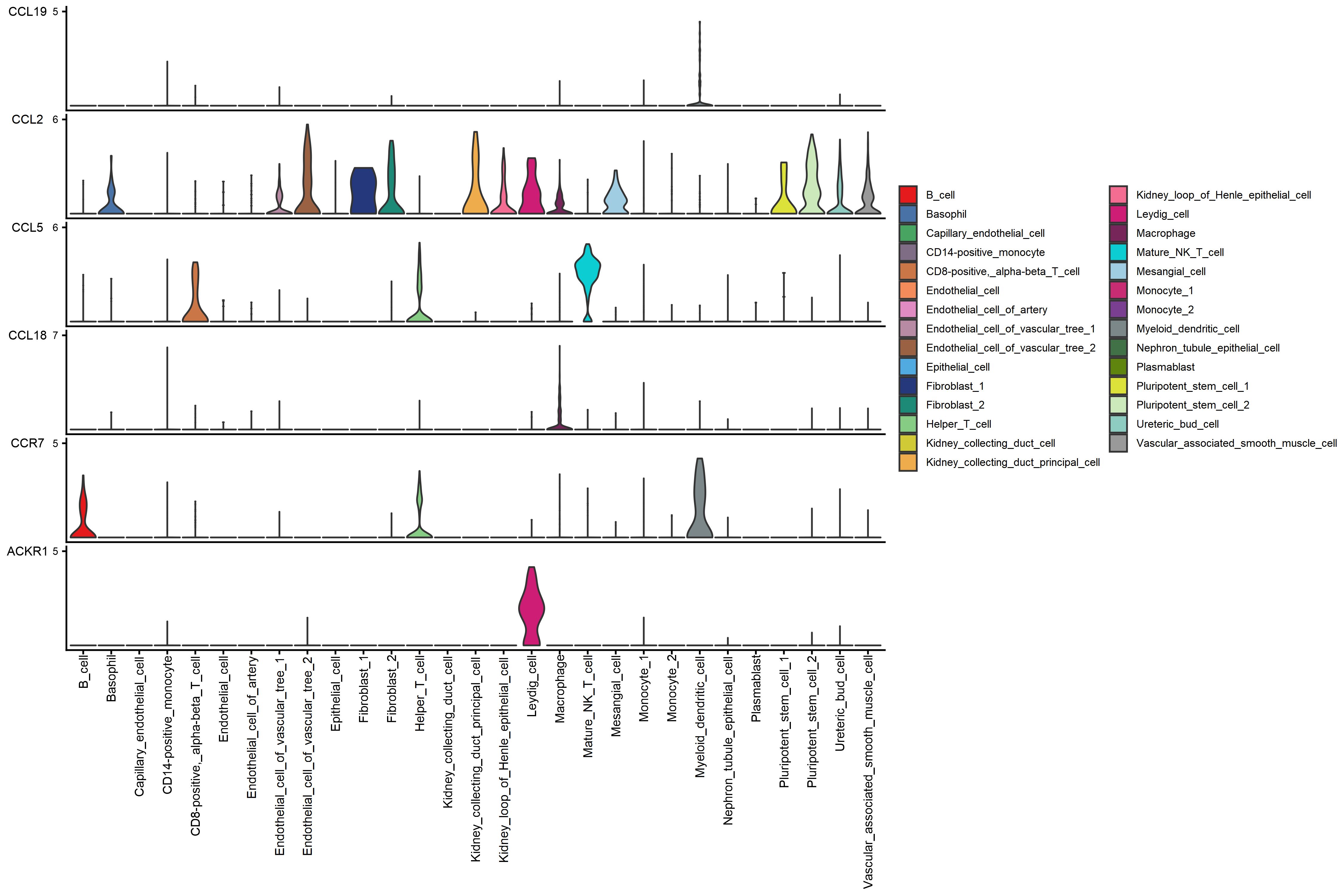
该通路受体配体在肿瘤和正常组织中的表达水平小提琴图
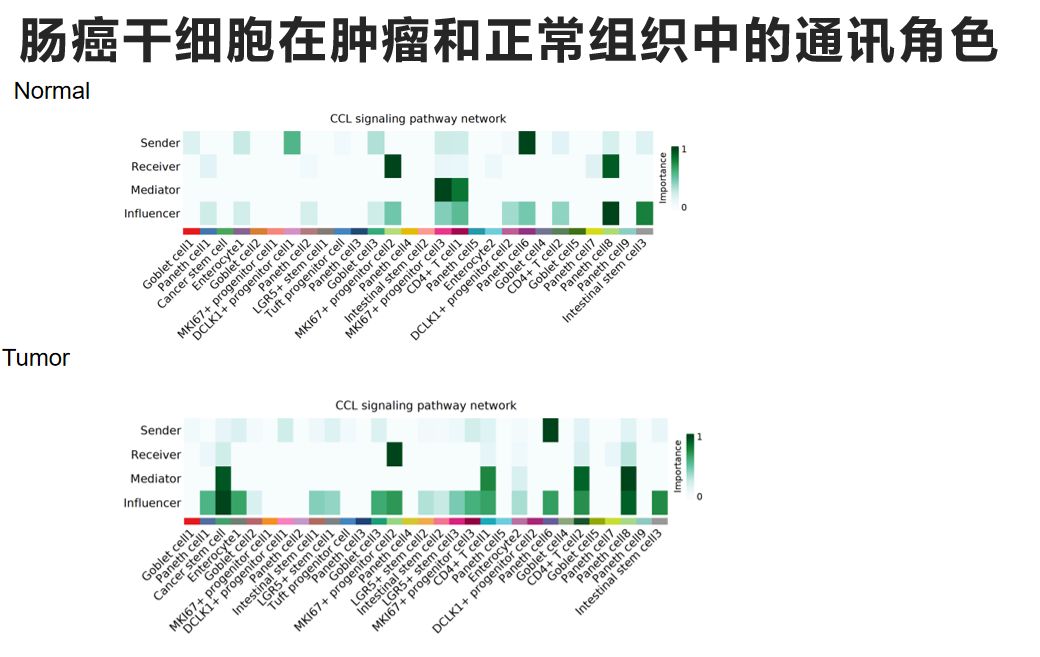
该模块结果中的子步骤cellchat6的结果图
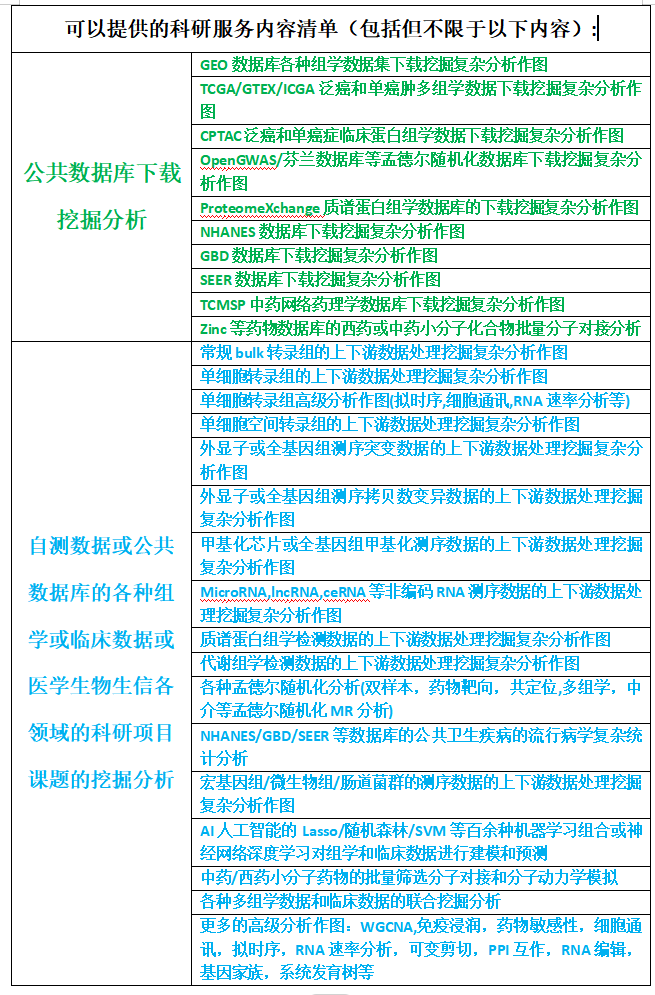
可以提供的科研服务清单