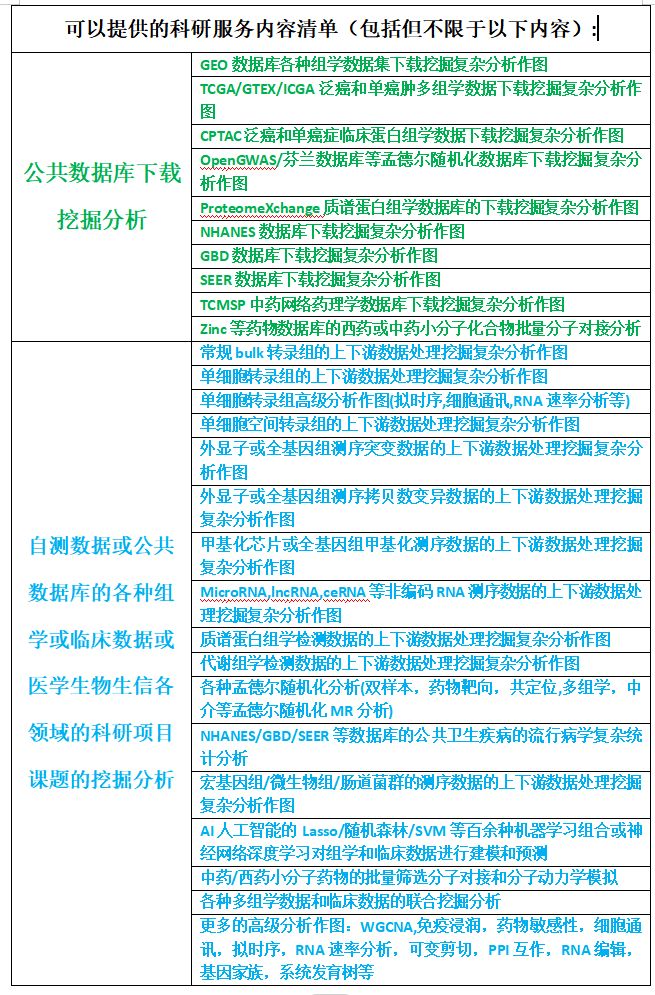
使用多种机器学习调参模型进行二分类建模的全流程教程,代做分析
使用多种机器学习调参模型进行二分类建模的全流程教程
机器学习全流程分析各个模块用到的总的参数文件
0. 分析参数文件
参数文件名称:total_analysis_params_demo.xlsx ,很多分析模块都是这个总的参数文件,我的这个总的参数文件如果有更新的话,我就上传到百度网盘。包括机器学习建模和可视化的所有分析模块都是用的这一个参数文件
从百度网盘下载该参数文件,该参数文件在百度网盘的位置:
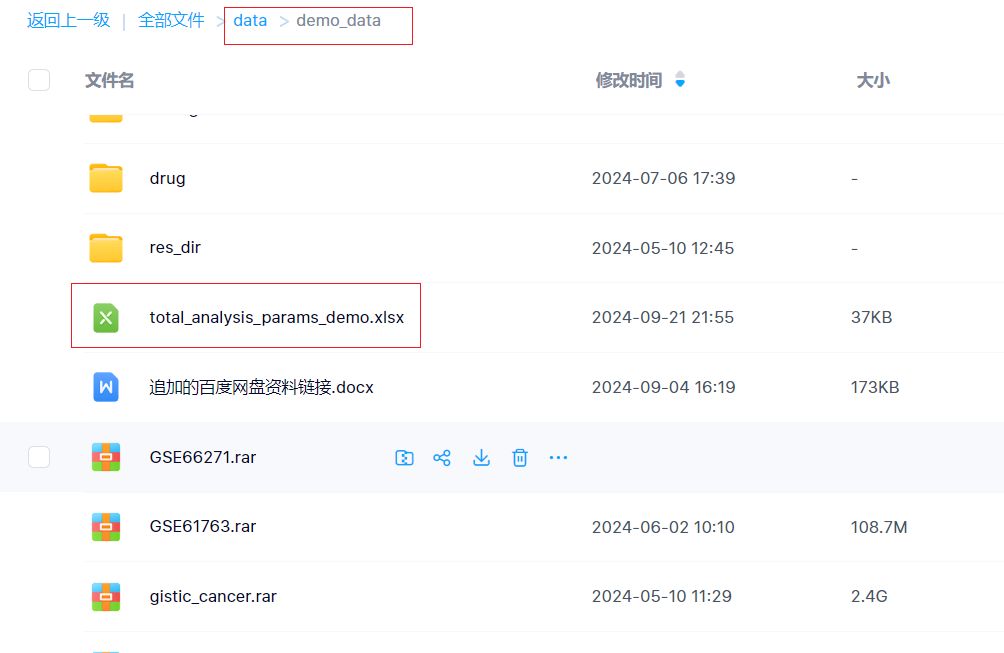
我们可以把这个参数文件下载到本地的:D:/omics_tools/demo_data这个目录下。下载后,在本地的文件为:
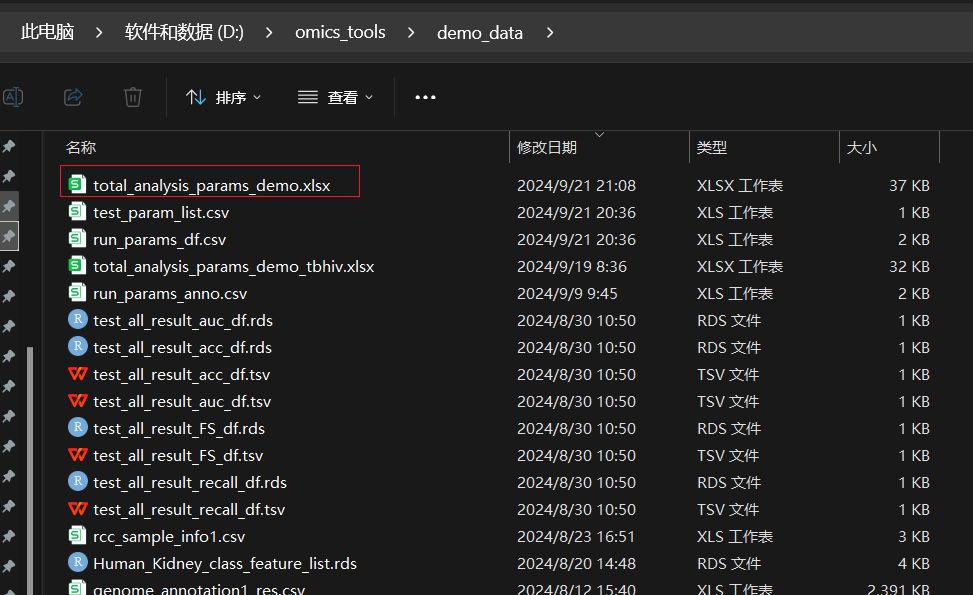
机器学习建模和可视化所有模块都用的这个总的参数文件的完整的文件路径就是:D:/omics_tools/demo_data/total_analysis_params_demo.xlsx
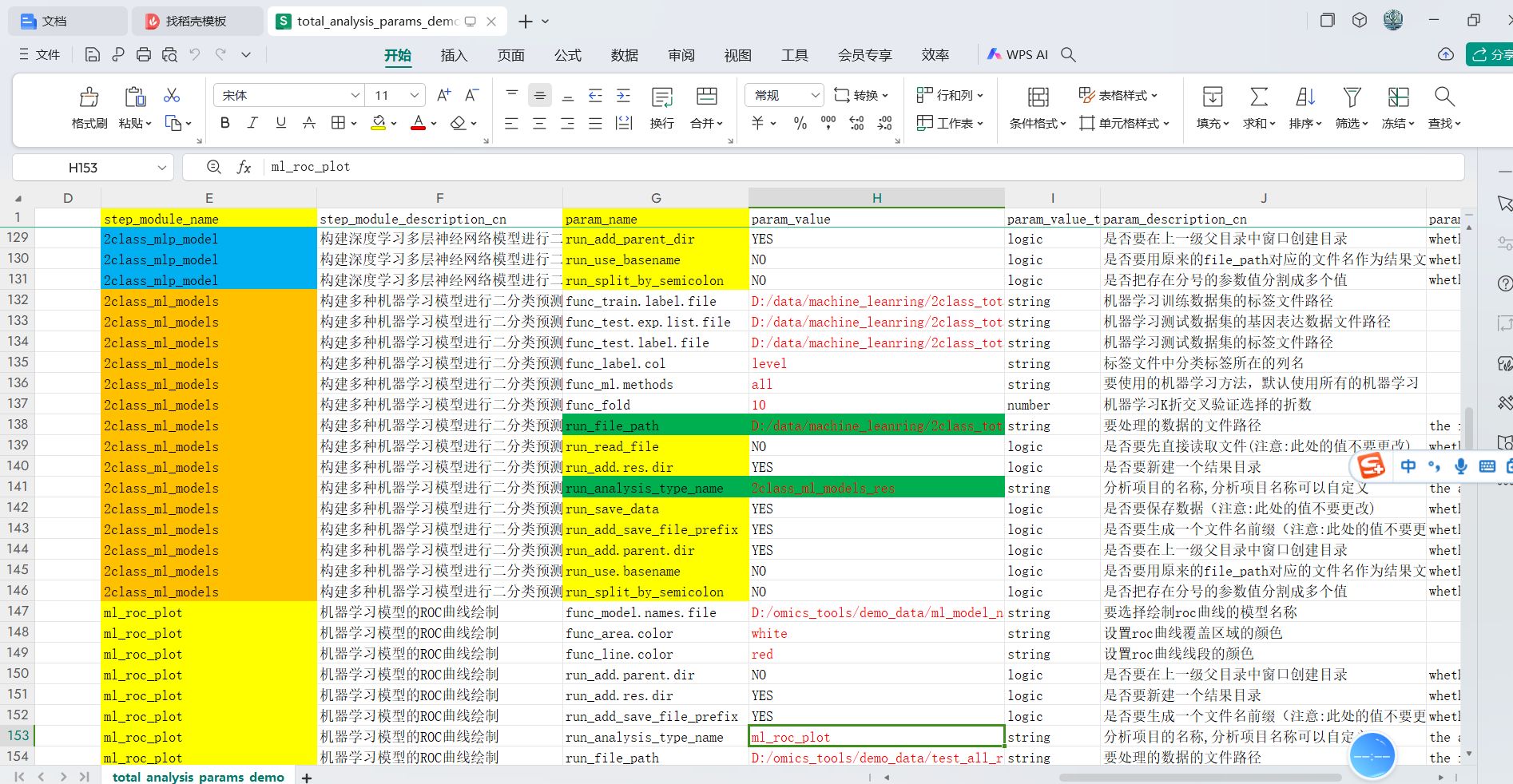
修改参数值的时候,只用改这个模块的参数值中我标红的那几个参数值,包括大家电脑中要分析的数据的文件路径和一些要调整和特别指定的重要参数值,没有标红的参数值,大家一律用默认的,不用修改。
机器学习的数据预处理和建模
对要进行机器学习或深度学习的数据进行整理和预处理
分析模块位置
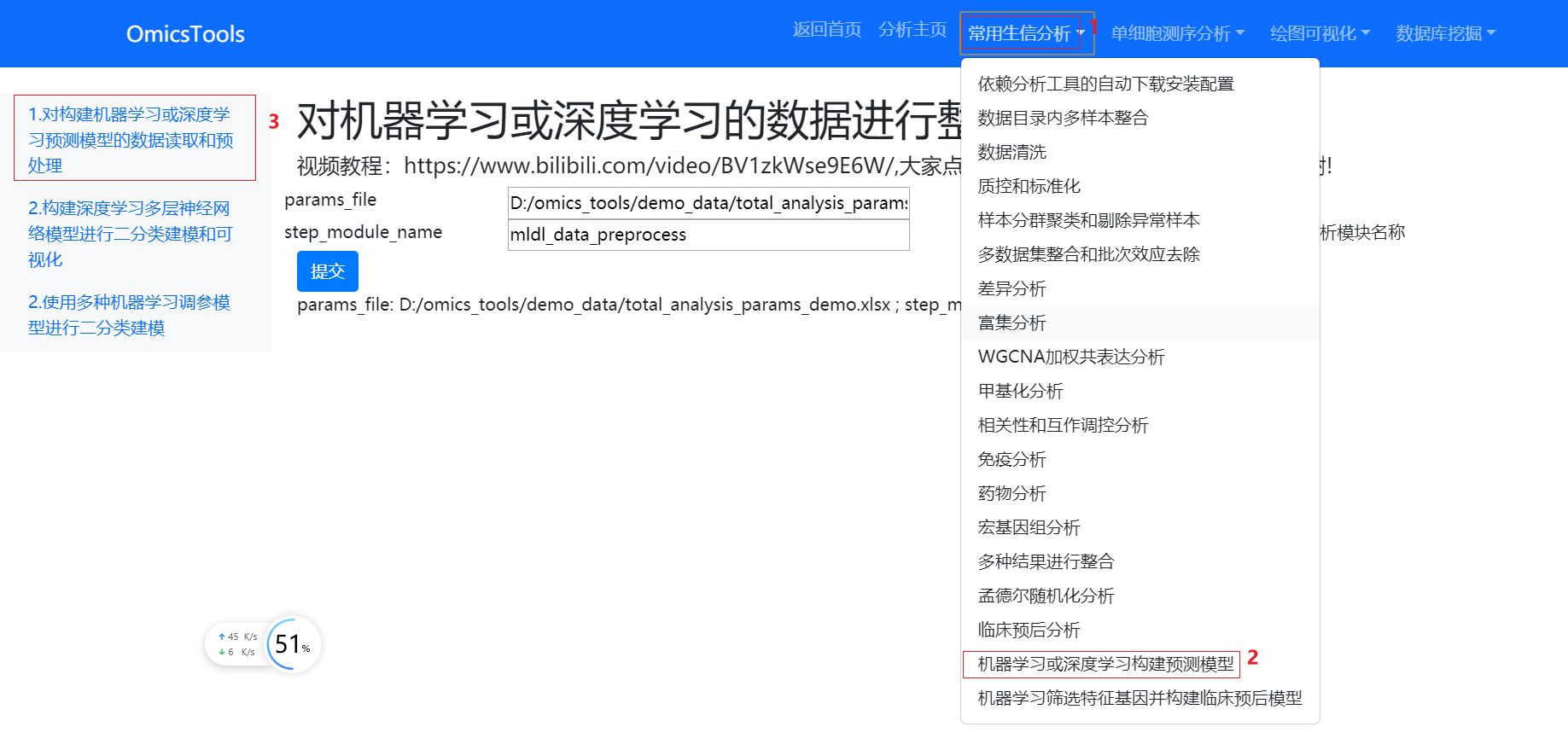
分析模块界面
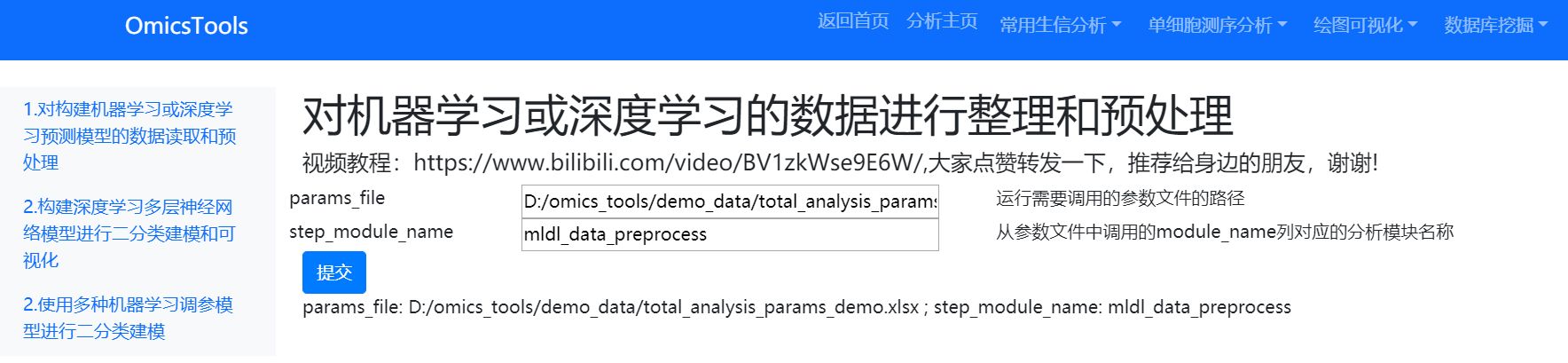
参数文件下载和使用
该模块的parms_file 需要给一个excel表的表格格式的参数文件,该参数文件可以在我的百度网盘中下载。
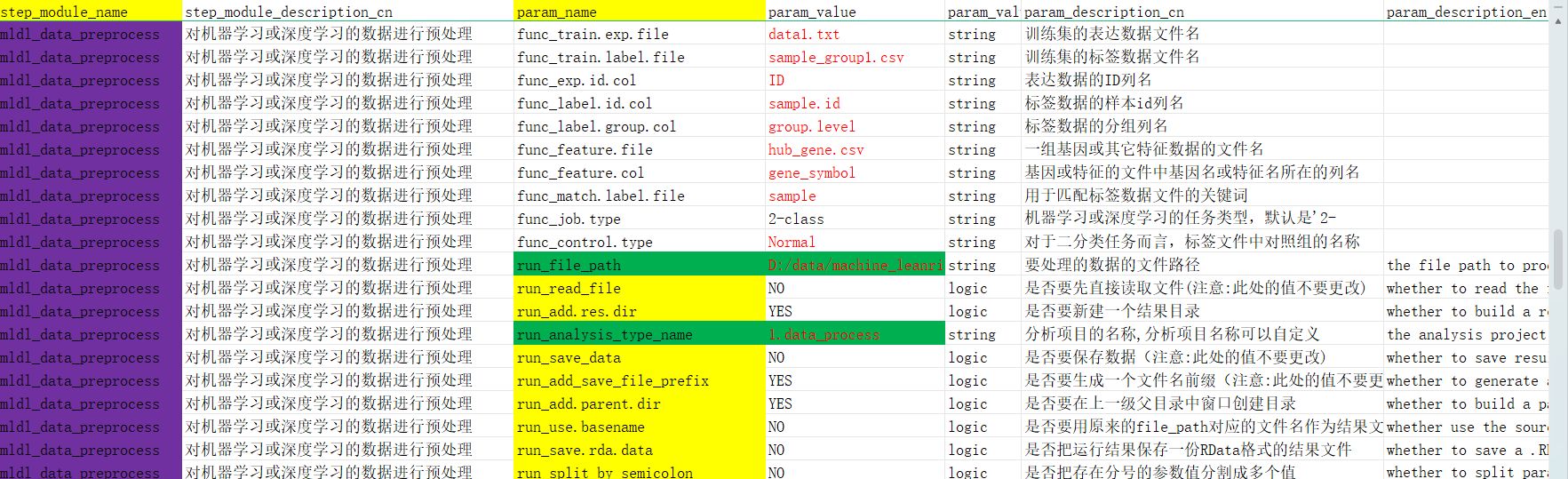
只需要在分析参数的这个xlsx的excel文件中修改一下mldl_data_preprocess分析模块下对应的在param_value列我标红的参数值,改成你自己的训练集和测试集文件所在的列名和文件中的基因id列名和样本id列名,样本分组列名即可,具体可以参考该模块下我的那个详细的教学视频,该视频观看链接已经放在了分析模块的内部了。
用到的训练集和测试集文件
文件目录下的所有文件
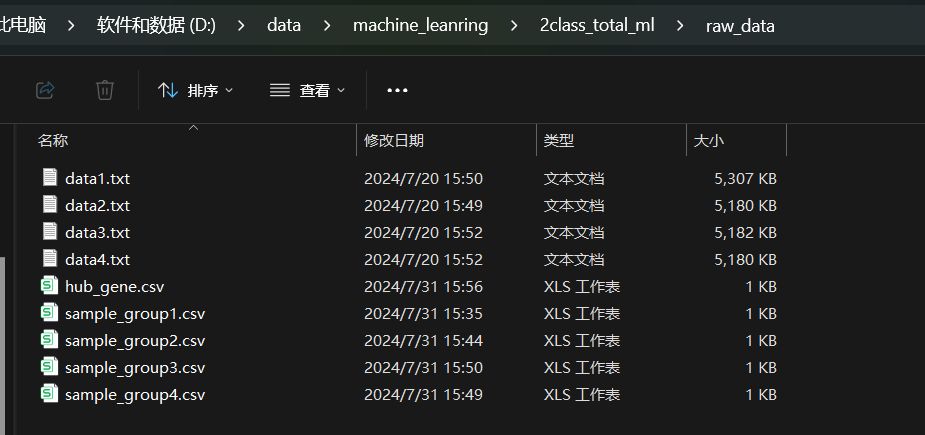
这是分析用到的所有数据文件存放的目录路径,其中:
- data1.txt是训练数据集文件,sample_group1.txt是该data1.txt对应的样本分组分类标签文件
- data2.txt, data3.txt, data4.txt是三个测试数据集文件,sample_group2.csv,sample_group3.csv,sample_group4.csv是这三个测试数据集分别对应的样本分组分类标签文件。当前这里是用了3个测试数据集文件和分类标签文件,大家也可以用更少或更多的测试数据集文件和分类标签文件
- hub_gene.csv 是机器学习或深度学习用到的一组关键基因列表,后面只会从data1.txt, data2.txt, data3.txt, data4.txt这些训练数据集和测试数据集中提取出这一组基因对应的表达数据进行建模和预测。
文件的具体内容
data1.txt, data2.txt, data3.txt, data4.txt这些基因表达数据集的文件内容
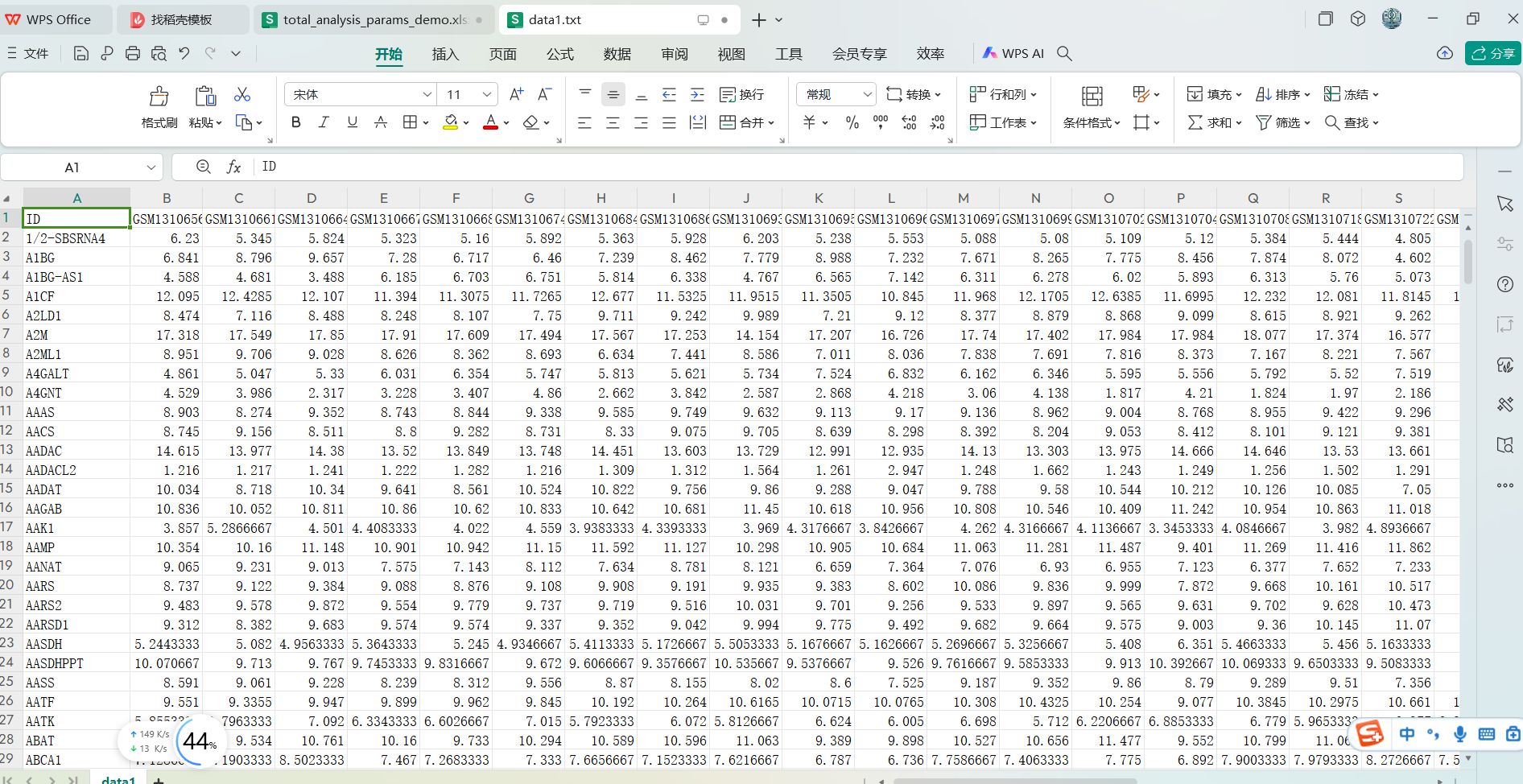
这些文件基本上都是第1列为基因名列,其它列都是样本列的这样的数据格式,即是一个行名为基因名,列名为样本编号的基因表达矩阵。
sample_group1.csv, sample_group2.csv,sample_group3.csv,sample_group4.csv对应的文件内容
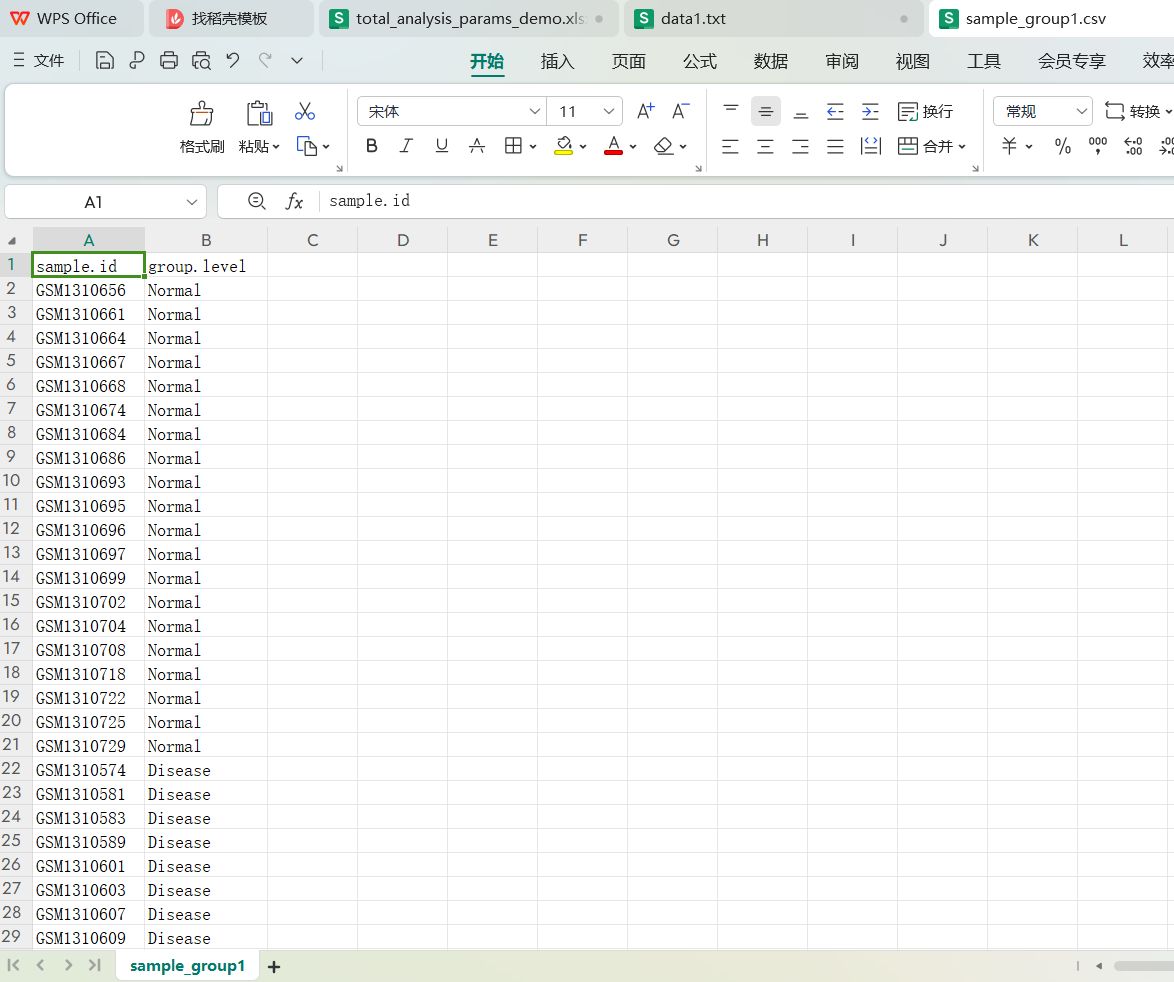
这些分类标签文件都只有两列,列名是sample.id和group.level, sample.id是样本id编号,group.level是样本的分类组名,这里的两个分类是Normal和Disease.
hub_gene.csv对应的文件内容
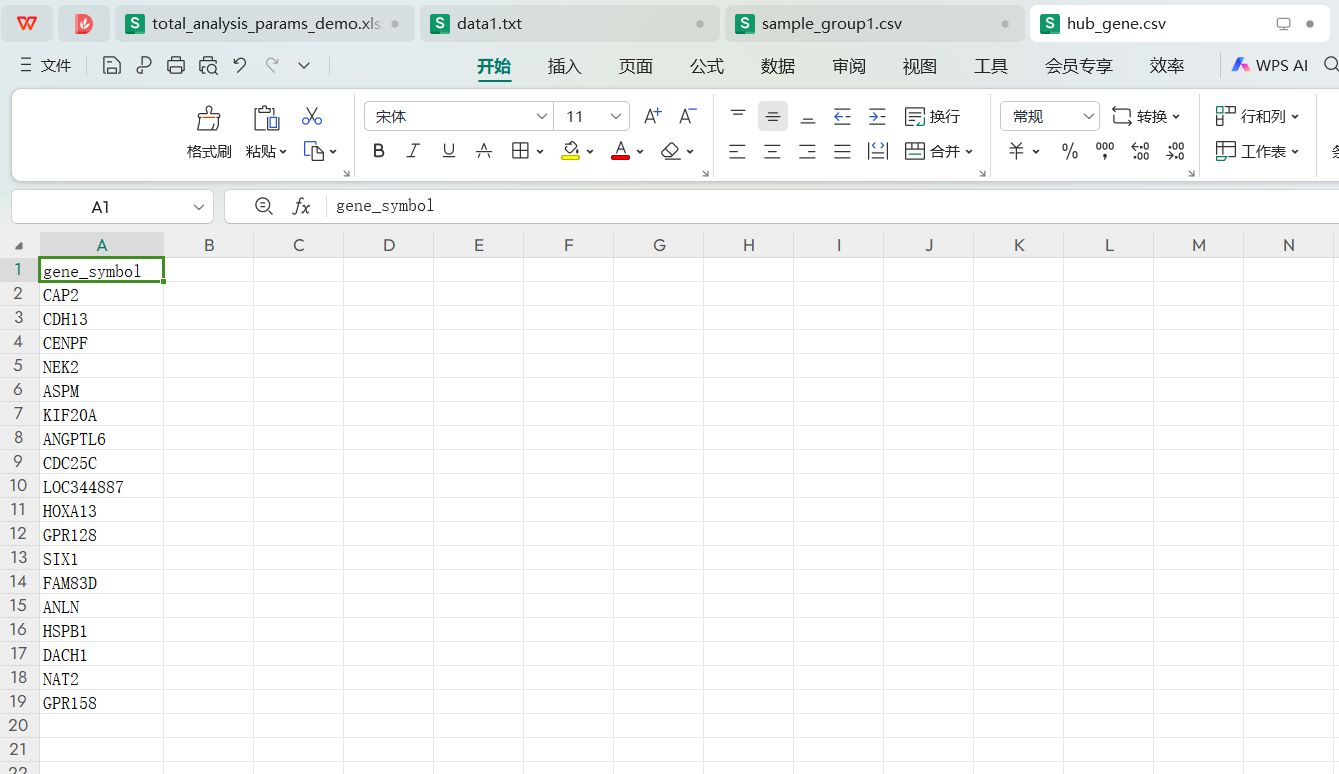
该文件中只有一组基因列表,列名是gene_symbol, 后续在分析中只会用到这一组基因进行数据处理,特征筛选和建模预测。
该分析模块的运行结果
分析界面执行完成的显示信息
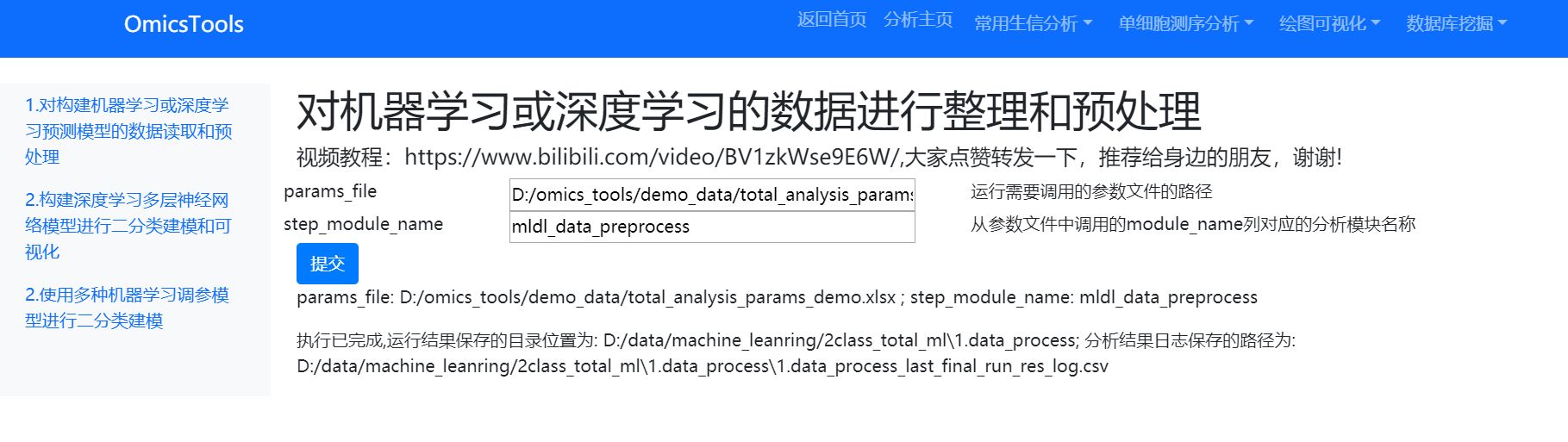
运行完成的结果目录
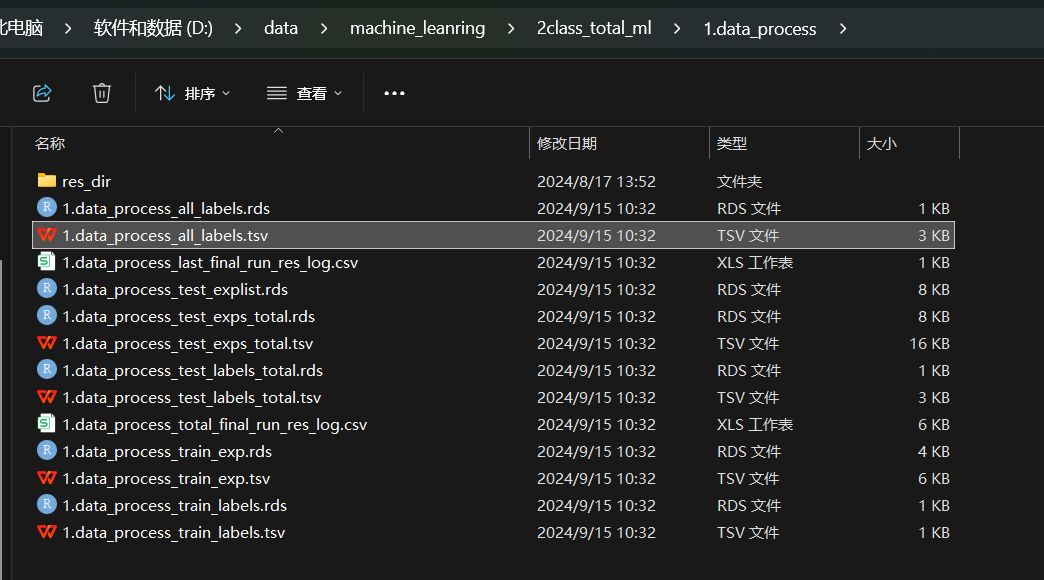
结果文件包括预处理好的测试集和训练集的表达数据集文件和分类标签文件,每个文件都有一份tsv文件和rds文件,tsv文件可以直接用excel打开,rds文件可以用Rstudio打开,二者的内容基本一致。
运行完成的文件内容
表达数据集的处理好的内容如下
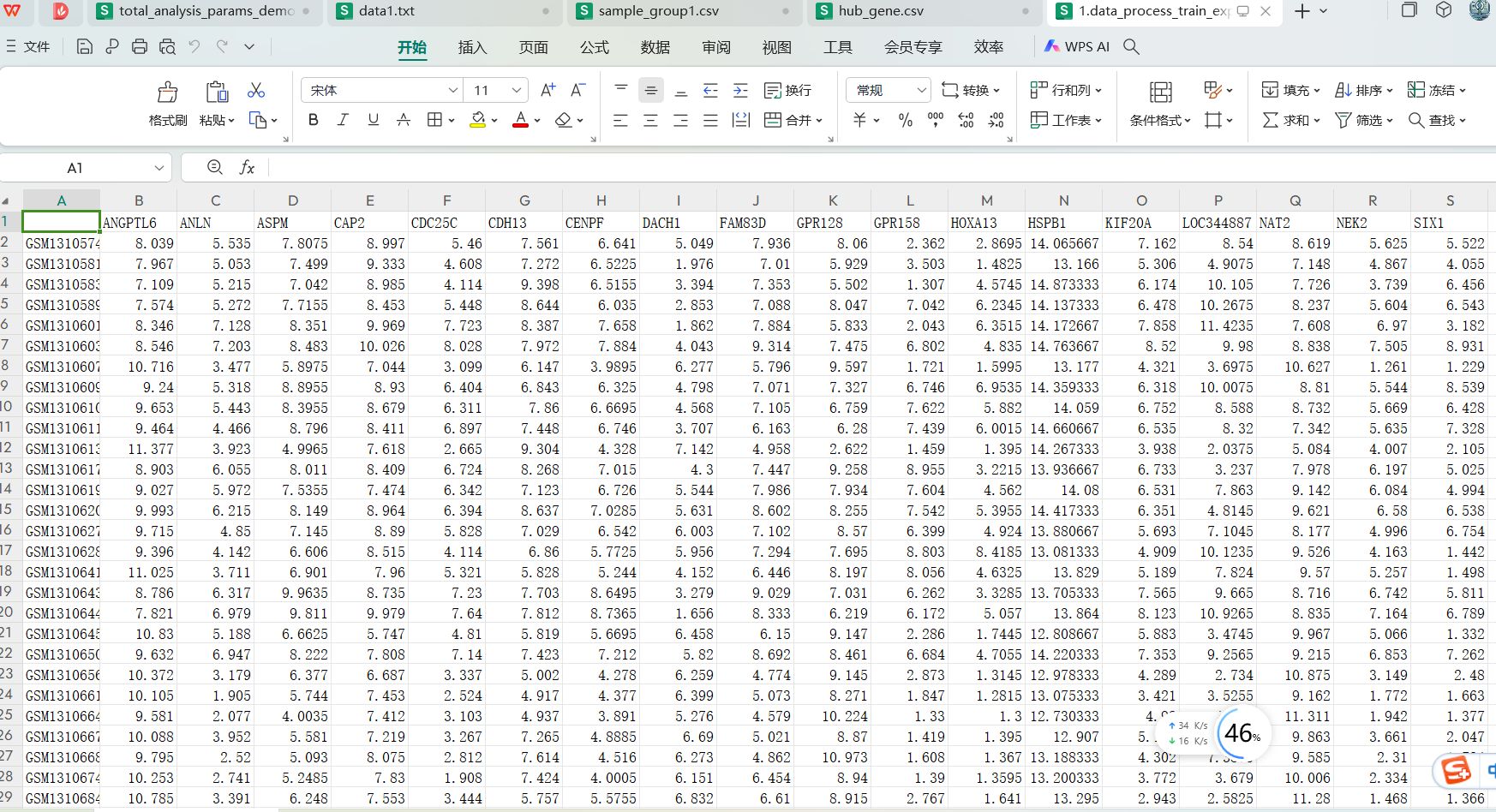
提取除了一组hub gene对应的表达矩阵,行名是样本编号,列名是这一组hub gene对应的表达数据
分组标签处理好的内容如下:
训练集标签文件处理好的内容:训练集标签文件data_process_train_labels.tsv处理好的内容如下:
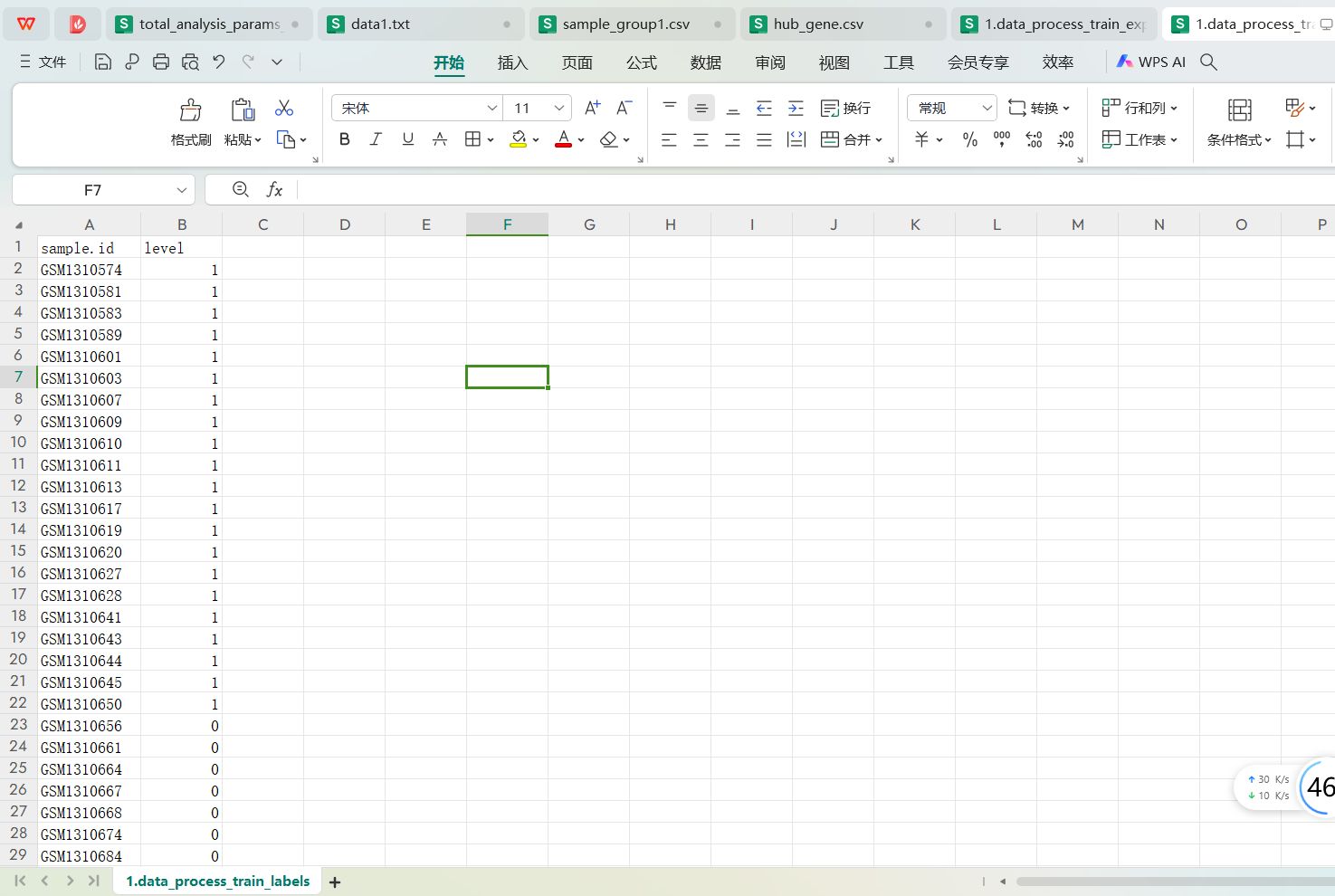
有两列数据,第一列是样本id,第二列是0,1分类标签,把原来的Normal变成了0, 把Disease变成了1
多个测试数据集的分类标签处理文件处理好的内容如下:1.data_process_test_labels_total.tsv文件内容:
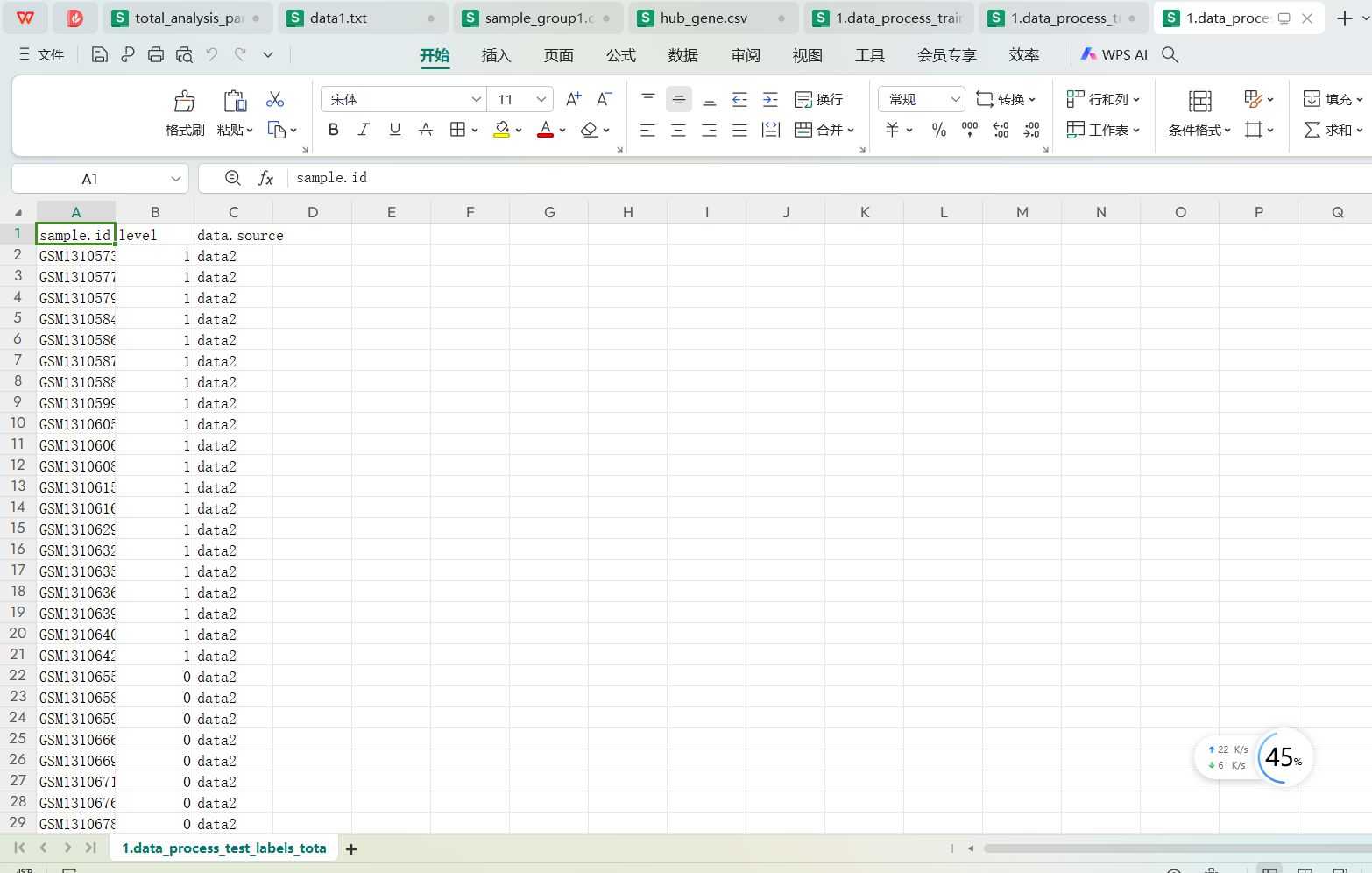
这里有三列数据,第一列是样本id,第二列是0,1分类标签,把原来的Normal变成了0, 把Disease变成了1,第三列是样本对应的数据集来源,因为这里用到了三个测试集,所有data.source列是data2,data3, data4这三个数据集的名称。
使用多种机器学习调参模型进行二分类建模
分析模块位置
软件界面
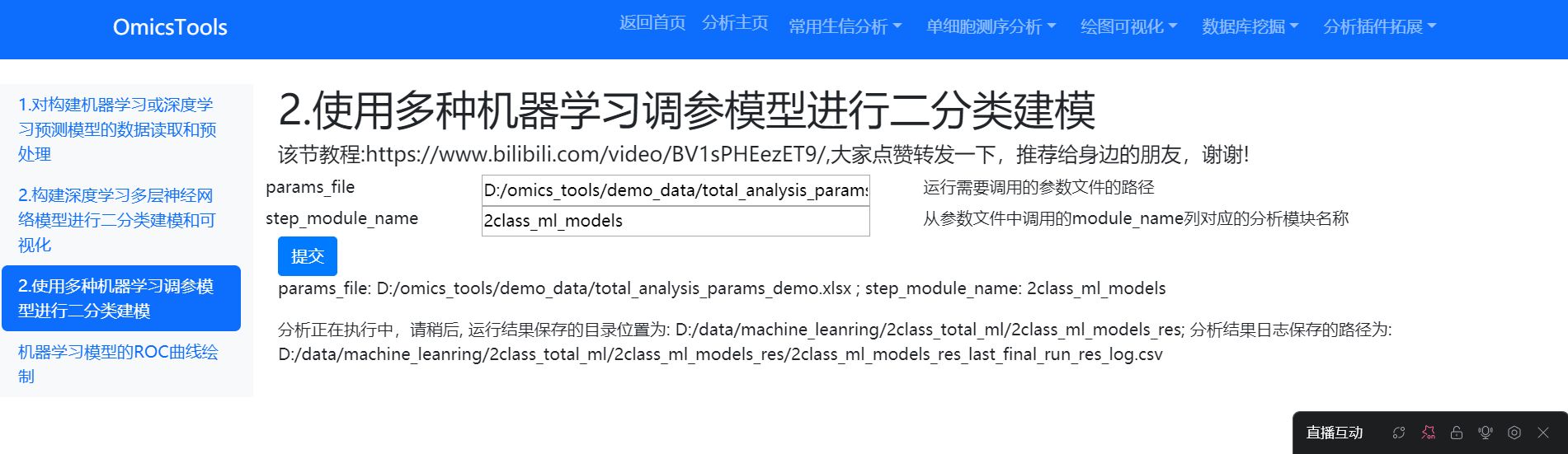
在软件界面里已经内置了该模块的适配教程链接,大家可以直接打开该b站课程网址来学习该模块的使用教程。
分析参数修改
所有机器学习系列模块用到的参数文件都是D:/omics_tools/demo_data/total_analysis_params_demo.xlsx这个参数文件,可以看我前面讲的直接从我百度网盘里把这个参数文件下载到D盘的omics_tools目录下的demo_data这个目录中。
在按照自己的数据修改分析参数进行自定义分析的时候,都是在total_analysis_params_demo.xlsx这个excel文件里进行修改参数值,修改参数值的时候,只用改这个模块的参数值中我标红的那几个参数值,包括大家电脑中要分析的数据的文件路径和一些要调整和特别指定的重要参数值,没有标红的参数值,大家一律用默认的,不用修改。
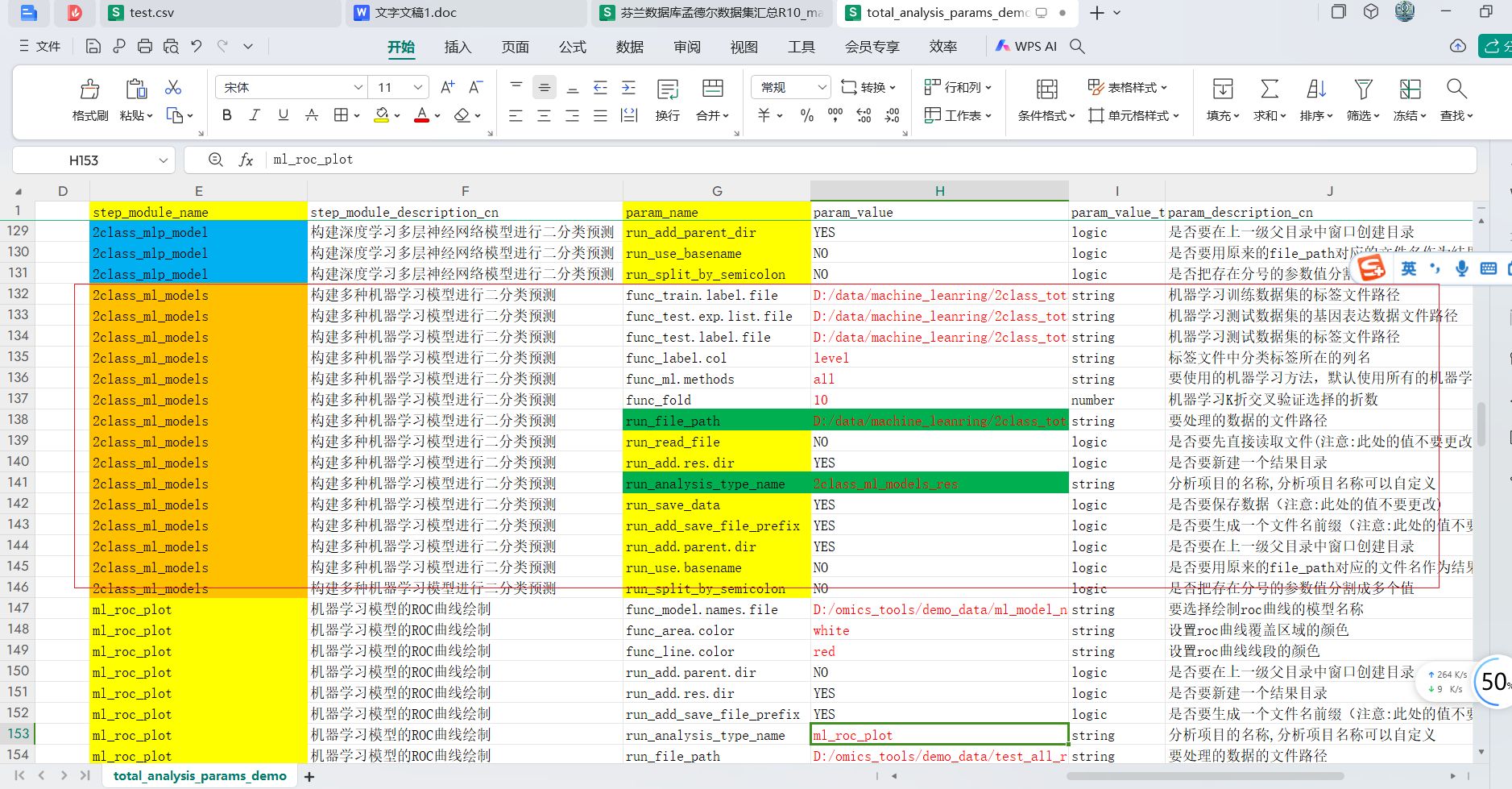
这个模块的step_module_name是2class_ml_models,参数值在参数文件中对应的信息为:
运行完成的结果
所有的结果文件列表
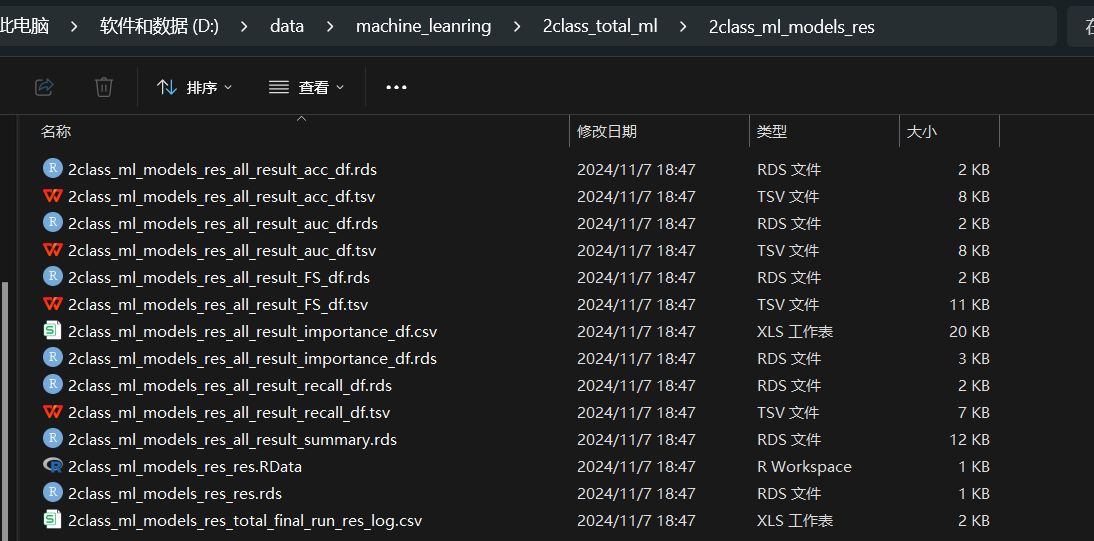
得到所有机器学习模型在多个测试集和训练集中的得分汇总表格:
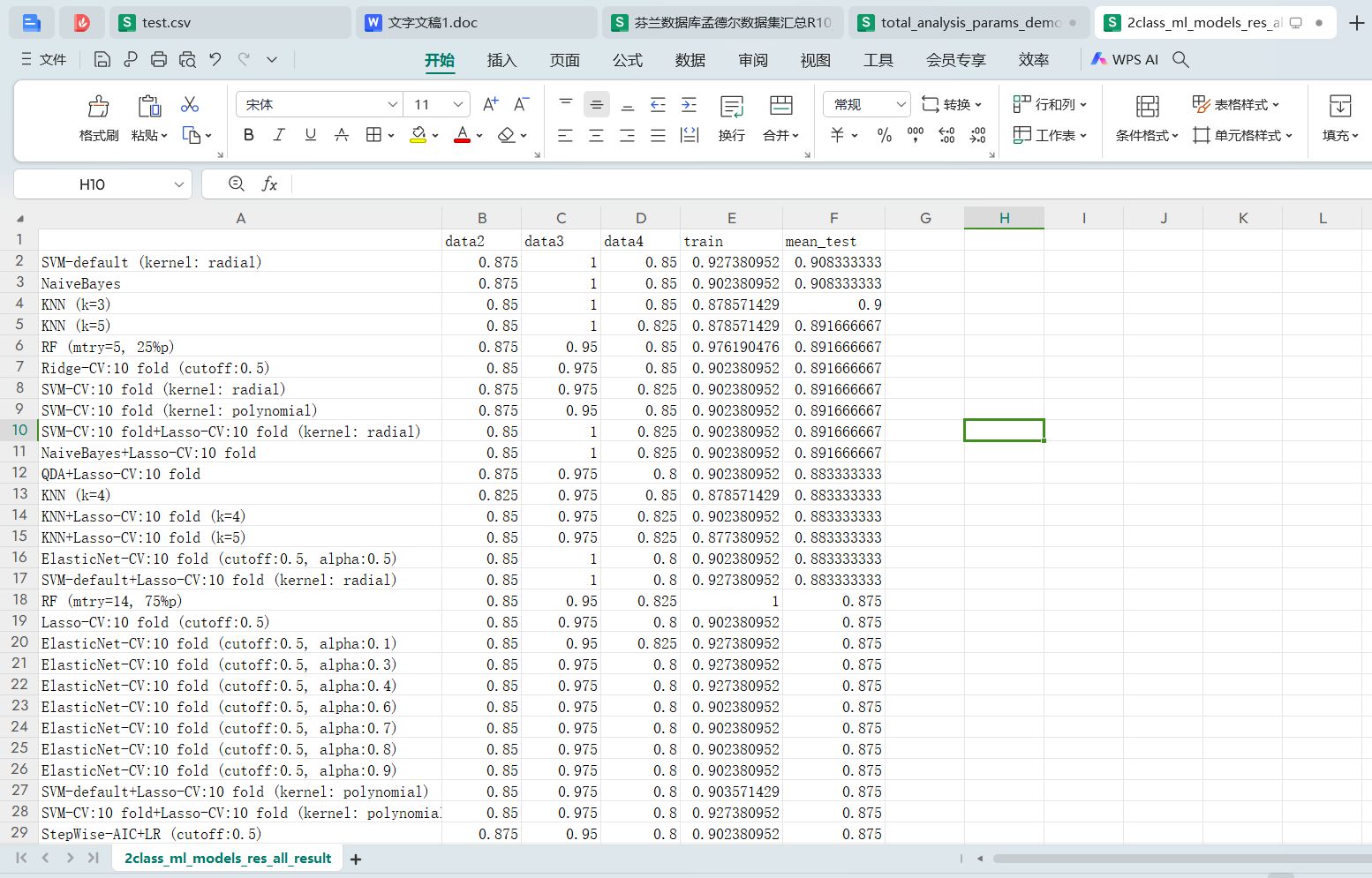
结果可视化和解读
按某个分组绘制水平分面条形图,展示多种机器学习的特征基因重要性
软件界面
在软件界面里已经内置了该模块的适配教程链接,大家可以直接打开该b站课程网址来学习该模块的使用教程。
分析参数修改
所有机器学习系列模块用到的参数文件都是D:/omics_tools/demo_data/total_analysis_params_demo.xlsx这个参数文件,可以看我前面讲的直接从我百度网盘里把这个参数文件下载到D盘的omics_tools目录下的demo_data这个目录中。
在按照自己的数据修改分析参数进行自定义分析的时候,都是在total_analysis_params_demo.xlsx这个excel文件里进行修改参数值,修改参数值的时候,只用改这个模块的参数值中我标红的那几个参数值,包括大家电脑中要分析的数据的文件路径和一些要调整和特别指定的重要参数值,没有标红的参数值,大家一律用默认的,不用修改。
这个模块的step_module_name是ml_gene_importance_barplot,参数值在参数文件中对应的信息为:
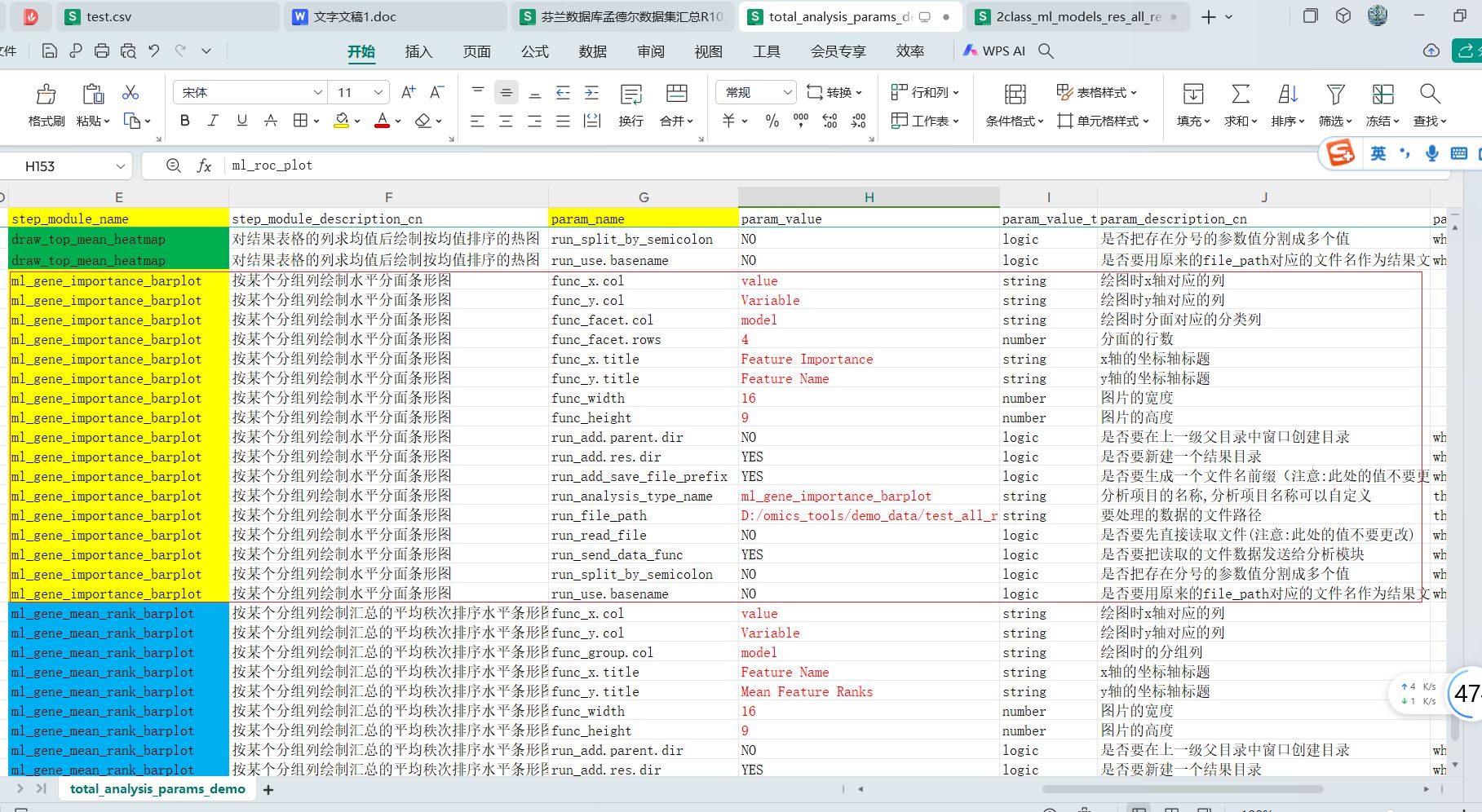
运行完成的结果
所有的结果文件列表
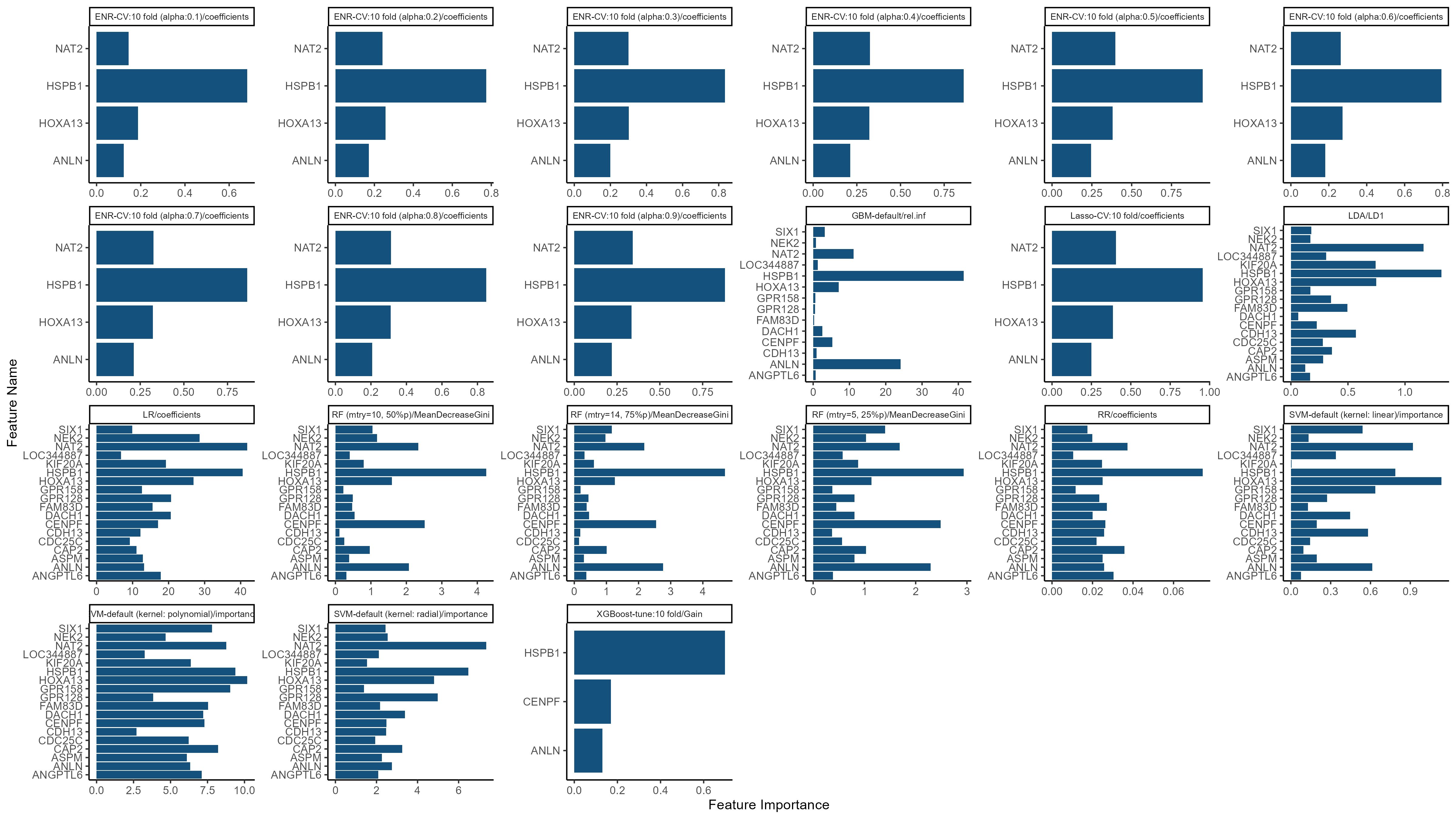
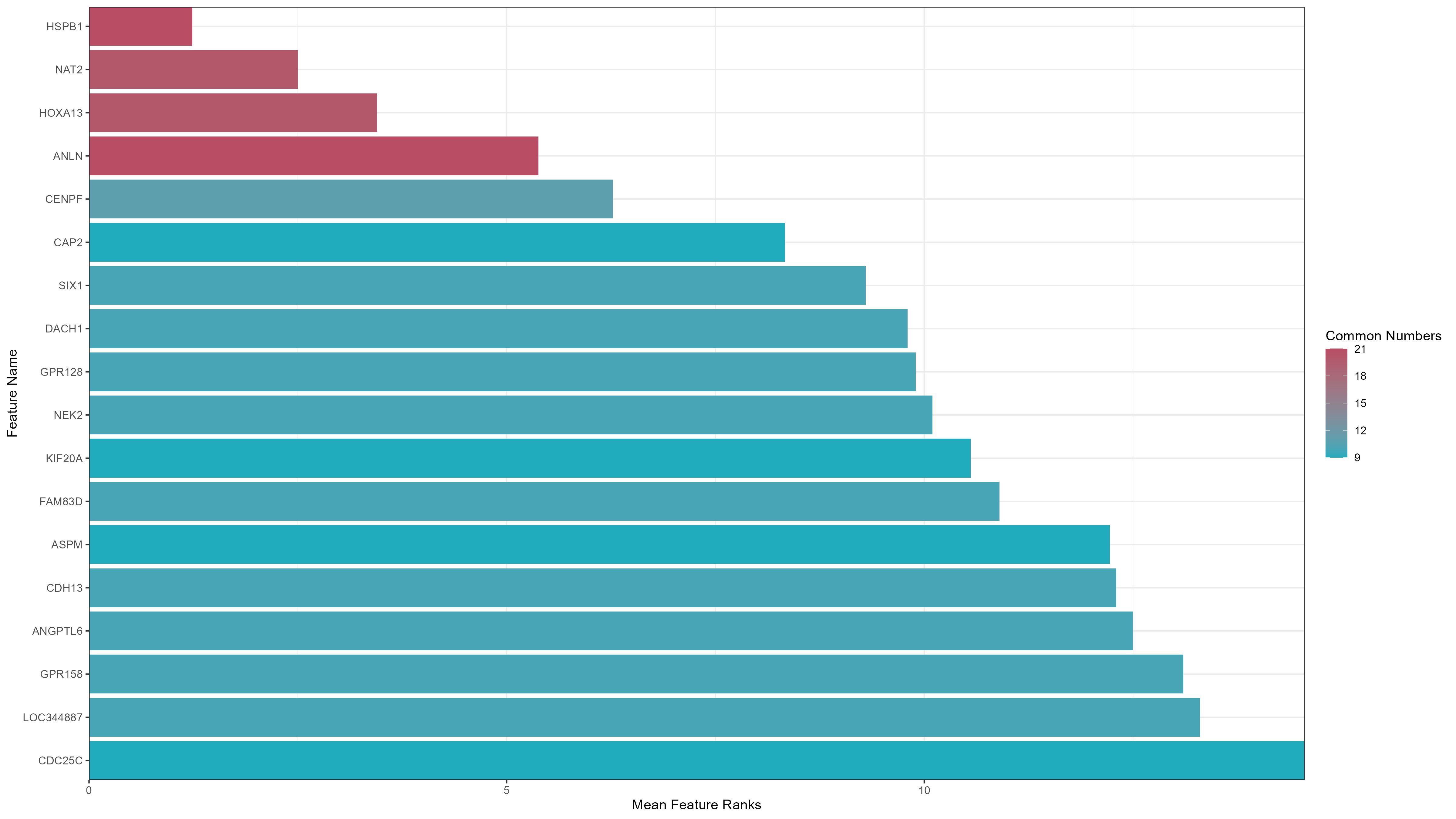
按某个分组列绘制汇总的平均秩次排序水平条形图,以机器学习的特征可视化
软件界面
分析参数修改
所有机器学习系列模块用到的参数文件都是D:/omics_tools/demo_data/total_analysis_params_demo.xlsx这个参数文件,可以看我前面讲的直接从我百度网盘里把这个参数文件下载到D盘的omics_tools目录下的demo_data这个目录中。
在按照自己的数据修改分析参数进行自定义分析的时候,都是在total_analysis_params_demo.xlsx这个excel文件里进行修改参数值,修改参数值的时候,只用改这个模块的参数值中我标红的那几个参数值,包括大家电脑中要分析的数据的文件路径和一些要调整和特别指定的重要参数值,没有标红的参数值,大家一律用默认的,不用修改。
这个模块的step_module_name是ml_gene_mean_rank_barplot,参数值在参数文件中对应的信息为:
运行完成的结果
所有的结果文件列表

ROC曲线的可视化和解读
ROC曲线的特异性和敏感性讲解
敏感性(Sensitivity)和特异性(Specificity)是二分类模型评价中非常重要的指标,它们用于衡量模型对正负样本的识别能力。我们逐一解读一下:
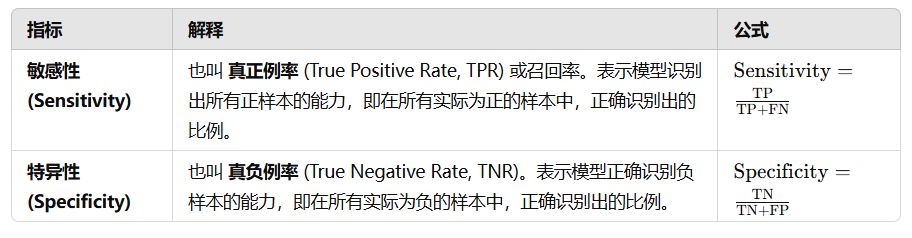
其中:
- TP(True Positive):模型将实际为正的样本预测为正。
- FN(False Negative):模型将实际为正的样本预测为负。
- TN(True Negative):模型将实际为负的样本预测为负。
- FP(False Positive):模型将实际为负的样本预测为正。
示例帮助理解:
假设我们在检测一种疾病时,用“是否患病”作为分类标准:
- 敏感性:指在所有真正患病的人中,模型检测出患病的比例。敏感性高的模型几乎不会漏掉真正患病的人。
- 特异性:指在所有未患病的人中,模型检测出未患病的比例。特异性高的模型几乎不会错判健康人患病。
进一步说明:
- 高敏感性:适合希望尽量减少漏诊的场景,比如疾病筛查,因为我们希望尽可能找出所有患病的人。
- 高特异性:适合希望尽量减少误诊的场景,比如监控系统的误报,因为我们希望系统只在真正有异常时报警。
总结
敏感性和特异性是两个相辅相成的指标。一般情况下,模型很难同时做到高敏感性和高特异性,需要根据实际需求进行权衡。
ROC曲线结果解读
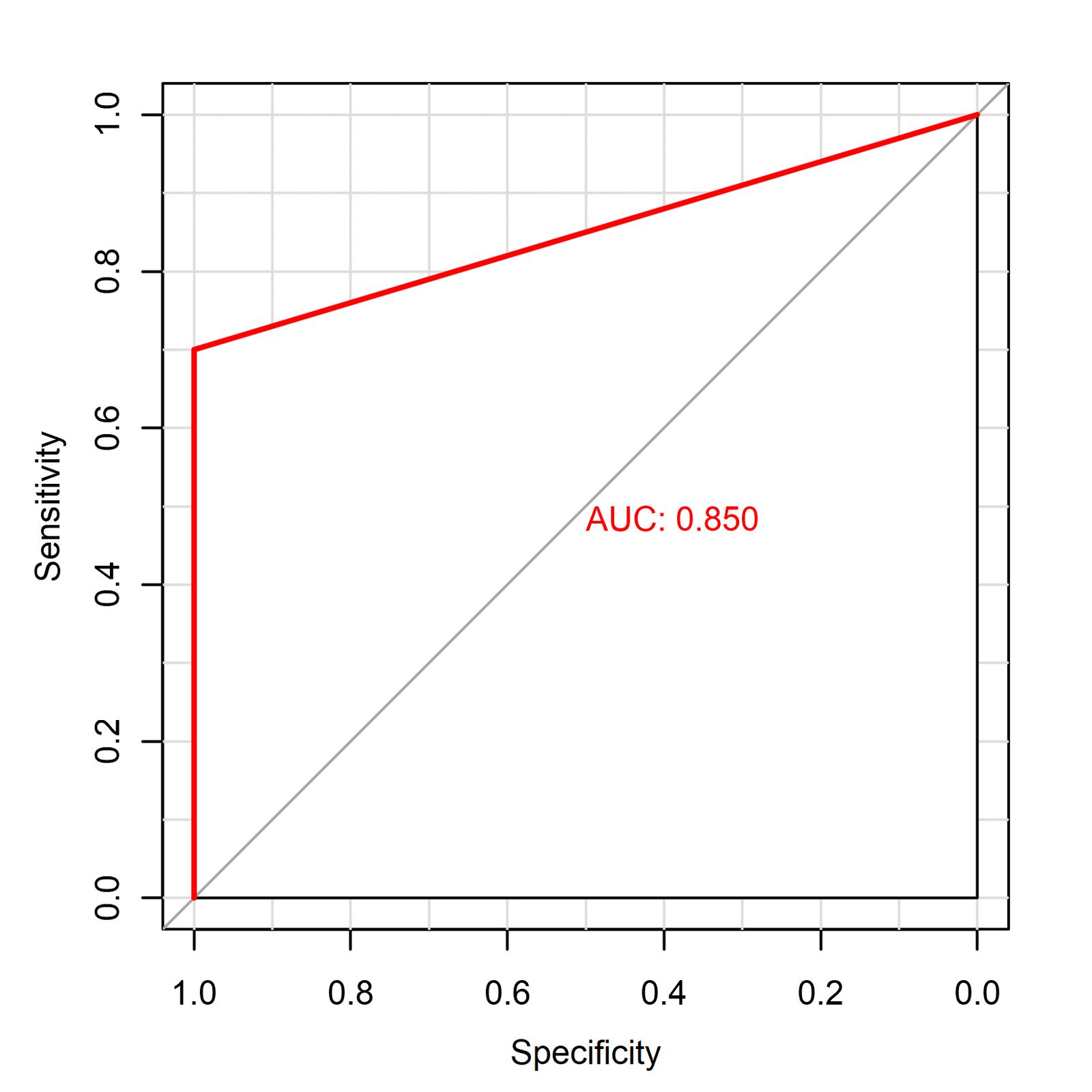
这个图展示的是一个ROC(Receiver Operating Characteristic)曲线,它用于评估二分类模型的性能。具体解读如下:
1. 横轴 (Specificity) 和纵轴 (Sensitivity):
- 横轴:表示 1 - 特异性(Specificity),也就是假阳性率 (FPR)。
- 纵轴:表示 敏感性(Sensitivity),即真正例率 (TPR)。
- 曲线从左下角 (0,0) 到右上角 (1,1) 逐渐变化,体现模型在不同分类阈值下的表现。
2. AUC(Area Under Curve)值:
- 图中标注了 AUC 值为 0.850。
- AUC 的值表示曲线下的面积,值的范围在 0.5 到 1 之间。值越接近 1,模型的分类效果越好。
- 0.850 代表模型的区分能力较好,但还不是完美模型。
3. 曲线解读:
- ROC 曲线的理想情况是尽可能靠近左上角(表示高敏感性和高特异性),这说明模型能够更好地区分正负样本。
- 你的曲线贴近左上角,但不完全紧贴,意味着模型在处理该分类问题时表现不错,但也有进一步提升的空间。
总结来看,这个模型在分类任务中具有良好的表现,能够比较准确地预测正负样本,但仍有一些改进余地。
有锯齿阶梯型的ROC曲线和更平滑的ROC曲线的区别和比较
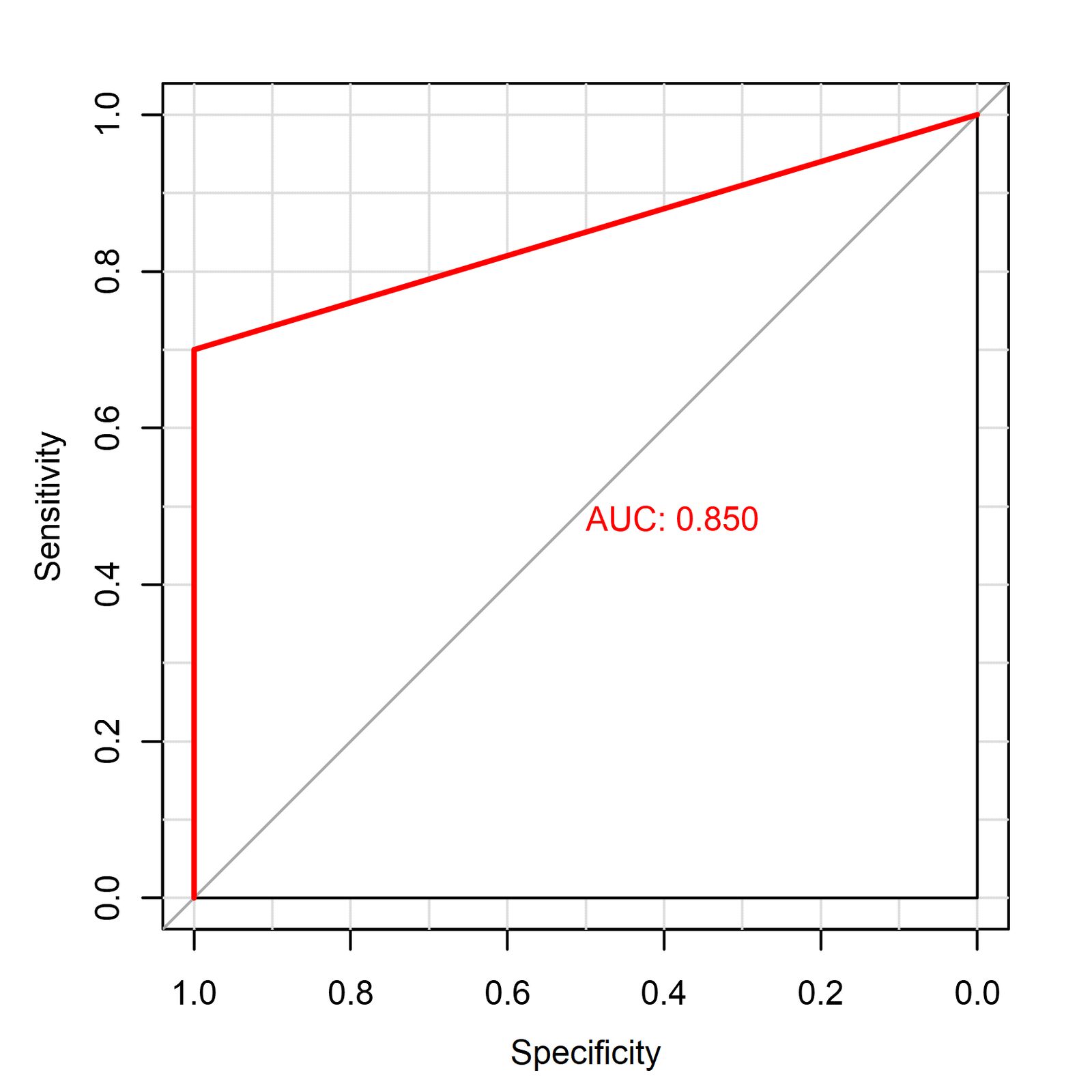
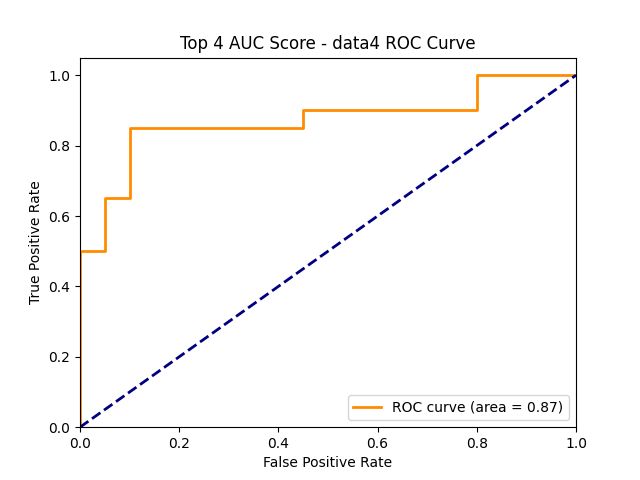
- 第一张图:曲线比较平滑,且一开始上升较快,在0.7之后逐渐趋向1。
- 第二张图:曲线有明显的阶梯状变化,表现出模型在某些区间的假阳性率上出现了跳跃。这可能意味着模型在这些特定区间上表现不够平滑或稳定。
- 对比结果:第一张图的曲线更加平滑,表明模型的预测可能更稳定;而第二张图在特定区间的阶梯状表现可能显示出该模型在某些决策阈值下的不连续性。
出现这种曲线形状差异的原因,通常与模型类型、数据集的特征、以及正负样本的分布密切相关。让我们来剖析原因,并探讨哪种性状的ROC曲线更优:
1. 原因分析
- 模型类型:一般来说,像K近邻(KNN)这类基于实例的模型,由于对每个样本的预测比较"硬"(硬分类),在ROC曲线上可能呈现出阶梯状(如第二张图),因为模型在某些决策阈值下无法灵活地调整预测概率。
- 数据特性:如果数据的正负样本分布不均匀,或者数据点非常有限(如小样本数据集),可能会导致ROC曲线呈现不规则的阶梯状。特别是在有限数据上训练的模型,可能在某些区域无法平滑地调整预测,造成ROC曲线的跳跃。
- 概率输出的平滑度:部分模型(如神经网络)输出的是连续概率,能够在不同的决策阈值下产生平滑的ROC曲线(如第一张图)。而另一类模型(如KNN)则可能输出离散分类值,因此ROC曲线会出现"分段"。
2. 哪种ROC曲线性状更好
- 平滑曲线(第一张图):这种性状一般出现在输出概率分布平滑的模型中,说明模型在不同的决策阈值下有较为稳定的表现。这种平滑性说明模型在各个阈值上的表现没有大的跳跃,通常能更好地捕捉样本的真实分布,是一种较为理想的ROC曲线性状。
- 阶梯状曲线(第二张图):这种性状并不一定差,但它往往意味着模型在特定阈值上表现得不够连续,可能在预测上存在波动。这种跳跃在一些特定场景下(如小数据集)可能是不可避免的,但一般来说,这种曲线反映了模型在某些区间内的预测能力不足。
总结
在大多数情况下,平滑的ROC曲线性状更好,因为它表示模型能够在各种决策阈值下稳定地处理正负样本分类。而阶梯状曲线往往说明模型在预测上不够连续,这可能带来不稳定的预测结果。